Vùng dị nhiễm sắc
Vùng dị nhiễm sắc là một dạng DNA được đóng gói rất chặt, gồm có nhiều loại. Những loại này nằm trải dài và nằm giữa hai thái cực của vùng dị nhiễm sắc là: vùng dị nhiễm sắc ổn định và không ổn định. Cả hai đều đóng một vai trò trong sự biểu hiện của gen. Vì vùng này được đóng gói chặt chẽ, nên cấu trúc này được cho là không thể tiếp cận với polymerase và do đó không được phiên mã, tuy nhiên theo Volpe et al. (2002),[1] và nhiều tài liệu khác sau đó,[2] phần lớn DNA này, trên thực tế, là có được phiên mã, nhưng nó liên tục được chuyển qua sự tắt phiên mã kích ứng bởi RNA (RITS hay RNA-induced transcriptional silencing). Các nghiên cứu gần đây với kính hiển vi điện tử và nhuộm OsO4 cho thấy rằng vùng đóng chặt không phải do chất nhiễm sắc.[3]
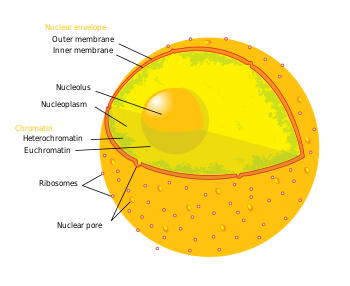
Vùng dị nhiễm sắc ổn định có thể ảnh hưởng đến các gen gần chính nó (biến đổi khảm do hiệu quả vị trí hay PEV). Nó có các trình tự lặp và hình thành các chức năng cấu trúc như tâm động hoặc telomere, ngoài việc hoạt động như một nơi thu hút các tín hiệu biểu hiện gen hoặc tín hiệu ức chế khác.
Vùng dị nhiễm sắc không ổn định là kết quả của các gen bị tắt thông qua nhiều cơ chế như khử acetyl hóa histone hoặc RNA tương tác Piwi (piRNA) thông qua RNAi. Nó không phải là trình tự lặp và cũng không có cấu trúc nhỏ gọn giống như vùng dị nhiễm sắc ổn định. Tuy nhiên, nhờ các tín hiệu phát triển hoặc môi trường cụ thể, nó có thể mất cấu trúc đóng xoắn của nó và có thể tham gia hoạt động phiên mã.[4]
Vùng dị nhiễm sắc có thể được liên hệ với di-và tri-methyl hóa H3K9 ở một số phần của bộ gen.[5]
Cấu trúc sửa
Chromatin được tìm thấy với hai dạng: nguyên nhiễm sắc và dị nhiễm sắc.[6] Ban đầu, hai dạng này được phân biệt về mặt tế bào học bằng cách so sánh chúng bắt màu mạnh như thế nào - vùng nguyên nhiễm sắc ít bắt màu, trong khi vùng dị nhiễm sắc lại bắt màu mạnh mẽ, cho thấy nó đóng gói chặt hơn. Các cùng dị nhiễm sắc thường được định vi ở ngoại vi của nhân. Mặc dù ta đã chia làm hai dạng đơn giản này, các bằng chứng gần đây ở cả hai đối tượng là động vật [7] và thực vật[8] đã gợi ý rằng có thể có nhiều hơn hai trạng thái mà ta nêu ra, và thực tế: nó có thể tồn tại đến bốn hay năm 'dạng', mỗi dạng có các dấu hiệu epigenetic khác nhau.
Vùng dị nhiễm sắc chủ yếu bao gồm các trình tự vệ tinh không hoạt động,[9] và nhiều gen bị ức chế ở các mức độ khác nhau, có cả một số gen không thể được biểu hiện kể cả khi có dạng nguyên nhiễm sắc.[10] Cả hai vị trí tâm động và telomere đều là dị nhiễm sắc, cũng giống như thể Barr-nhiễm sắc thể X thứ hai bị bất hoạt ở phụ nữ.
Chức năng sửa
Vùng dị nhiễm sắc có liên quan đến một số chức năng, từ điều hòa gen đến việc bảo vệ tính toàn vẹn của nhiễm sắc thể;[11] một số vai trò này có thể là do cấu trúc DNA bị đóng gói rất chặt, làm cho nó ít tiếp cận hơn với các yếu tố protein thường gắn với DNA hoặc các yếu tố đi kèm. Ví dụ, nếu đầu tận cùng chuỗi DNA mà lộ ra thì có thể được hiểu bởi tế bào rằng DNA bị hư hỏng hoặc của virus, kích hoạt dừng chu kỳ tế bào, sửa chữa DNA hoặc phá hủy phần đó đi, chẳng hạn như bởi các endonuclease trong vi khuẩn.
Một số vùng nhiễm sắc được đóng gói rất chặt, tương đương với mức độ cuộn xoắn của nhiễm sắc thể trong phân bào. Vùng dị nhiễm sắc thường được di truyền theo dòng; khi một tế bào phân chia, hai tế bào con thường chứa vùng dị nhiễm sắc trong cùng một vùng DNA, dẫn đến di truyền biểu sinh. Các biến thể có thể làm cho vùng dị nhiễm sắc xâm lấn các gen lân cận hoặc rời xa các gen ở các cực của các miền. Các yếu tố phiên mã được có thể bị ức chế do vị trí (bằng cis) tại các miền ranh giới này. Điều này làm tăng mức biểu hiện thay đổi từ tế bào này sang tế bào khác,[12] có thể được chứng minh bằng biến đổi khảm do hiệu quả vị trí.[13] Các trình tự cách li có thể hoạt động như một rào cản trong những trường hợp hiếm hoi mà vùng dị nhiễm sắc ổn định và các gen có biểu hiện cao được đặt cạnh nhau (ví dụ trình tự cách li 5'HS4 ngược dòng locus β-globin gà,[14] và loci trong hai Saccharomyces spp.[15][16]).
Chú thích sửa
- ^ Volpe, Thomas A.; Kidner, Catherine; Hall, Ira M.; Teng, Grace; Grewal, Shiv I. S.; Martienssen, Robert A. (ngày 13 tháng 9 năm 2002). “Regulation of heterochromatic silencing and histone H3 lysine-9 methylation by RNAi”. Science. 297 (5588): 1833–1837. doi:10.1126/science.1074973. ISSN 1095-9203. PMID 12193640.
- ^ “What is the current evidence showing active transcription withinin...”. www.researchgate.net. Truy cập ngày 30 tháng 4 năm 2016.
- ^ Ou, Horng D.; Phan, Sébastien; Deerinck, Thomas J.; Thor, Andrea; Ellisman, Mark H.; O’Shea, Clodagh C. (ngày 28 tháng 7 năm 2017). “ChromEMT: Visualizing 3D chromatin structure and compaction in interphase and mitotic cells”. Science (bằng tiếng Anh). 357 (6349): eaag0025. doi:10.1126/science.aag0025. ISSN 0036-8075. PMC 5646685. PMID 28751582.
- ^ Oberdoerffer, P; Sinclair, D (2007). “The role of nuclear architecture in genomic instability and ageing”. Nature Reviews Molecular Cell Biology. 8: 692–702. doi:10.1038/nrm2238.
- ^ Rosenfeld, Jeffrey A; Wang, Zhibin; Schones, Dustin; Zhao, Keji; Desalle, Rob; Zhang, Michael Q (ngày 31 tháng 3 năm 2009). “Determination of enriched histone modifications in non-genic portions of the human genome”. BMC Genomics. 10 (1): 143. doi:10.1186/1471-2164-10-143. PMC 2667539. PMID 19335899.
- ^ Elgin, S.C. (1996). “Heterochromatin and gene regulation in Drosophila”. Current Opinion in Genetics & Development. 6 (2): 193–202. doi:10.1016/S0959-437X(96)80050-5. ISSN 0959-437X.
- ^ van Steensel, B. (2011). “Chromatin: constructing the big picture”. The EMBO Journal. 30 (10): 1885–95. doi:10.1038/emboj.2011.135. PMC 3098493. PMID 21527910.
- ^ Roudier, François; và đồng nghiệp (2011). “Integrative epigenomic mapping defines four main chromatin states in Arabidopsis”. The EMBO Journal. 30 (10): 1928–1938. doi:10.1038/emboj.2011.103. PMC 3098477. PMID 21487388.
- ^ Lohe, A.R.; và đồng nghiệp (ngày 1 tháng 8 năm 1993). “Mapping Simple Repeated DNA Sequences in Heterochromatin of Drosophila Melanogaster”. Genetics. 134 (4): 1149–74. ISSN 0016-6731. PMC 1205583. PMID 8375654.
- ^ Lu, B.Y.; và đồng nghiệp (ngày 1 tháng 6 năm 2000). “Heterochromatin protein 1 is required for the normal expression of two heterochromatin genes in Drosophila”. Genetics. 155 (2): 699–708. ISSN 0016-6731. PMC 1461102. PMID 10835392.
- ^ Grewal SI, Jia S (tháng 1 năm 2007). “Heterochromatin revisited”. Nature Reviews Genetics. 8 (1): 35–. doi:10.1038/nrg2008. PMID 17173056. Bản gốc lưu trữ 24 Tháng 2 2017. Truy cập ngày 18 tháng 9 năm 2013.
An up-to-date account of the current understanding of repetitive DNA, which usually doesn't contain genetic information. If evolution makes sense only in the context of the regulatory control of genes, we propose that heterochromatin, which is the main form of chromatin in higher eukaryotes, is positioned to be a deeply effective target for evolutionary change. Future investigations into assembly, maintenance and the many other functions of heterochromatin will shed light on the processes of gene and chromosome regulation.
Chú thích có tham số trống không rõ:|quotes=(trợ giúp); Kiểm tra giá trị ngày tháng trong:|archive-date=(trợ giúp) - ^ Fisher, Amanda G.; Matthias Merkenschlager (tháng 4 năm 2002). “Gene silencing, cell fate and nuclear organisation”. Current Opinion in Genetics & Development. 12 (2): 193–7. doi:10.1016/S0959-437X(02)00286-1. ISSN 0959-437X. PMID 11893493.
- ^ Zhimulev, I.F.; và đồng nghiệp (tháng 12 năm 1986). “Cytogenetic and molecular aspects of position effect variegation in Drosophila melanogaster”. Chromosoma. 94 (6): 492–504. doi:10.1007/BF00292759. ISSN 1432-0886.Quản lý CS1: nhiều tên: danh sách tác giả (liên kết)
- ^ Burgess-Beusse, B; và đồng nghiệp (tháng 12 năm 2002). “The insulation of genes from external enhancers and silencing chromatin”. Proc. Natl. Acad. Sci. USA. 9 (Suppl 4): 16433–7. doi:10.1073/pnas.162342499. PMC 139905. PMID 12154228.
- ^ Noma, K.; và đồng nghiệp (tháng 8 năm 2001). “transitions in distinct histone H3 methylation patterns at the heterochromatin domain boundaries”. Science. 293 (5532): 1150–5. doi:10.1126/science.1064150. PMID 11498594.
- ^ Donze, D.; R.T. Kamakaka (2000). “RNA polymerase III and RNA polymerase II promoter complexes are heterochromatin barriers in Saccharomyces cerevisiae”. The EMBO Journal. 20 (3): 520–31. doi:10.1093/emboj/20.3.520. PMC 133458. PMID 11157758.