Công nghệ nano DNA
Công nghệ nano DNA là việc thiết kế và chế tạo cấu trúc acid nucleic nhân tạo cho mục đích công nghệ. Trong lĩnh vực này, axit nucleic được sử dụng làm vật liệu kỹ thuật phi sinh học cho công nghệ nano thay vì truyền tải thông tin di truyền trong tế bào sống. Các nhà nghiên cứu trong lĩnh vực này đã chế tạo thành công những cấu trúc tĩnh tại như cấu trúc tinh thể 2 chiều và 3 chiều, ống nano, khối đa diện, và các hình dạng tùy ý, cũng như những thiết bị hoạt động được như các cỗ máy phân tử và máy tính DNA. Lĩnh vực này đã bắt đầu được ứng dụng như một công cụ để giải đáp những vấn đề của khoa học cơ bản như trong sinh học cấu trúc và lý sinh học, bao gồm các ứng dụng trong các ngành tinh thể học tia X và phổ học cho việc xác định cấu trúc protein. Các ứng dụng khác trong ngành điện tử kích thước phân tử và y học nano cũng đang được nghiên cứu.
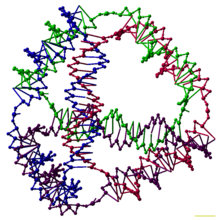
Nền tảng khái niệm cho công nghệ nano được xây dựng bởi Nadrian Seeman đầu những năm 1980, nhưng lĩnh vực này chỉ bắt đầu thu hút sự quan tâm rộng rãi từ giữa thập niên 2000. Việc sử dụng các axit nucleic dựa trên quy tắc ghép cặp base chặt chẽ của chúng, cho phép chỉ những đoạn trên dải DNA với trình tự chuỗi bổ sung cho nhau gắn kết để tạo thành cấu trúc chuỗi xoắn kép bền chặt. Điều này cho phép thiết kế trình tự chuỗi theo ý đồ để tổng hợp một cách lọc lựa DNA thành những cấu trúc đích phức tạp với cấu trúc bậc nano được kiểm soát chính xác. Có một số phương pháp tổng hợp khác nhau cho mục đích này, bao gồm các cấu trúc xếp gạch tổng hợp những cấu trúc nhỏ hơn, các cấu trúc gấp lại sử dụng phương pháp DNA origami, và các cấu trúc có thể tái định hình sử dụng các kỹ thuật dịch chuyển dải. Trong khi tên của lĩnh vực này nhắc tới DNA, những nguyên lý tương tự cũng đã được áp dụng với các axit nucleic khác, khiến cho đôi khi người ta gọi nó bằng tên khác là công nghệ nano axit nucleic.
Các khái niệm cơ bản
sửaTính chất của axit nucleic
sửaCông nghệ nano thường được định nghĩa là ngành nghiên cứu các vật liệu và thiết bị với những yếu tố nhỏ hơn 100 nanomet. Cụ thể công nghệ nano DNA là một ví dụ của tự tổng hợp phân tử từ dưới lên (bottom-up), trong đó các thành phần nguyên tử tổ chức thành các cấu trúc bền vững một cách tự phát; dạng cụ thể của những cấu trúc này do các tính chất vật lý và hóa học của các thành phần mà người thiết kế chọn quyết định.[4] Trong công nghệ nano DNA, các vật liệu thành phần là các dải axit nucleic như DNA; các dải này thường được tổng hợp và luôn được sử dụng ngoài môi trường tế bào sống. DNA rất phù hợp cho việc xây dựng cấu trúc nano bởi vì liên kết giữa hai dải axit nucleic phụ thuộc vào các quy luật ghép cặp base đơn giản mà con người đã hiểu rõ, và tạo nên những cấu trúc nano xác định từ cấu trúc xoắn kép. Những tính chất này khiến cho việc tổng hợp cấu trúc axit nucleic dễ dàng kiểm soát thông qua thiết kế chuỗi. Đặc tính này vắng mặt trong các vật liệu khác trong công nghệ nano, bao gồm protein, vốn rất khó thiết kế, và các hạt nano, vốn không có khả năng tự tổng hợp.[5]
Cấu trúc của một phân tử axit nucleic bao gồm một chuỗi các nucleotide phân biệt bởi nucleobase mà chúng chứa đựng. Trong DNA, 4 base hiện diện là adenine (A), cytosine (C), guanine (G), và thymine (T). Axit nucleic có một thuộc tính đặc biệt, đó là hai phân tử chỉ liên kết với nhau tạo thành một chuỗi xoắn kép nếu hai chuỗi là bổ sung cho nhau, nghĩa là chúng tạo nên các chuỗi phù hợp theo cặp base, theo nguyên tắc A chỉ liên kết với T, và C chỉ với G.[5][6] Bởi vì sự hình thành các cặp base phù hợp là thuận lợi về mặt nhiệt động (năng lượng tự do của hệ giảm), trong hầu hết mọi trường hợp các dải axit nucleic sẽ liên kết với nhau theo cách tối đa hóa số base ghép cặp phù hợp. Trình tự base trong một hệ các dải do đó sẽ quyết định mô hình liên kết và cấu trúc tổng thể theo một cách có thể kiểm soát dễ dàng. Trong công nghệ nano DNA, trình tự base của các dải được thiết kế theo ý đồ của nhà nghiên cứu sao cho tương tác ghép cặp base khiến cho các dải tổng hợp thành hình thể mong muốn.[3][5] Trong khi DNA là vật liệu chủ yếu được dùng, các cấu trúc bao gồm các axit nucleic khác như RNA và axit peptit nucleic (APN) cũng đã được chế tạo.[7][8]
Các lĩnh vực con
sửaCông nghệ nano DNA đôi khi phân thành hai nhánh nghiên cứu chồng lấn lên nhau: công nghệ nano DNA cấu trúc và công nghệ nano DNA động lực. Công nghệ DNA cấu trúc (tiếng Anh: Structural DNA nanotechnology, đôi khi viết tắt là SDN), tập trung vào việc tổng hợp và phân tích các vật liệu và phức hợp từ axit nucleic tổng hợp nên một trạng thái cuối tĩnh, cân bằng. Trong khi đó, công nghệ nano DNA động lực xét tới các phức hợp với những hành vi trong trạng thái không cân bằng hữu ích như khả tăng tái cấu trúc dựa trên một kích thích vật lý hoặc hóa học. Những phức hợp như vậy, chẳng hạn các thiết bị cơ học nano axit nucleic, kết hợp các đặc điểm của cả hai lĩnh vực con cấu trúc và động lực.[9][10]
Các cấu trúc được kiến tạo bởi công nghệ nano DNA cấu trúc sử dụng các cấu trúc axit nucleic phân nhánh topo chứa các mối nối. (Ngược lại, hầu hết các DNA trong sinh học tồn tại như những chuỗi xoắn kép không phân nhánh.) Một trong những cấu trúc phân nhánh đơn giản nhất là một mối nối bốn nhánh bao gồm 4 dải DNA đơn lẻ, từng phần của chúng bổ sung cho nhau theo một kiểu mẫu nhất định. Không giống như trong mối nối Holliday, mỗi nhánh của mối nối nhân tạo bất động này có một trình tự base khác nhau, khiến cho điểm nối cố định ở một vị trí nhất định. Nhiều mối nối có thể kết hợp vào cùng phức hợp, chẳng hạn như trong môtip xuyên chéo kép (tiếng Anh: double-crossover, DX), bao gồm hai miền xoắn kép song song với những dải riêng lẻ vắt qua giữa các miền ở hai điểm xuyên chéo. Về mặt topo mỗi điểm xuyên chéo tự nó là một mối nối bốn nhánh, nhưng bị giới hạn ở một định hướng duy nhất, trái với mối nối bốn nhánh đơn linh hoạt, cung cấp sự bền chắc khiến cho motip DX thích hợp làm một khối xây dựng nên những phức hợp DNA lớn hơn.[3][5]
Công nghệ nano DNA sử dụng một cơ chế gọi là dịch chuyển dải can thiệp chỗ đứng chân cho phép các phức hợp axit nucleic tái cấu trúc để đạp lại sự thêm vào một dải axit nucleic mới. Trong phản ứng này, dải mới tới liên kết với một miền chỗ đứng chân một dải của một phức hợp hai dải, và sau đó chuyển một trong các dải trong phức hợp ban đầu thông qua một quá trình di chuyển nhánh. Hệ quả chung cục là một trong các dải bị thay thế bởi một dải khác.[9] Ngoài ra, các cấu trúc và thiết bị tự tái cấu trúc có thể chế tạo từ các axit nucleic chức năng như deoxyribozyme và ribozyme, vốn có khả năng thực hiện các phản ứng hóa học, và các aptamer có thể liên kết với những protein đặc biệt hoặc những phân tử nhỏ.[11]
Công nghệ nano DNA cấu trúc
sửaCông nghệ nano DNA cấu trúc, đôi khi gọi tắt là SDN, tập trung vào việc tổng hợp và phân tích các vật liệu và phức hợp axit nucleic mà sự tổng hợp có một điểm mút ổn định, cân bằng. Cấu trúc xoắn kép của axit nucleic có dạng hình học 3 chiều vững chắc, xác định, cho phép tiên đoán và thiết kế các cấu trúc của những phức hợp axit nucleic phức tạp hơn. Nhiều cấu trúc như vậy đã được tạo ra, bao gồm các cấu trúc 2 và 3 chiều, các cấu trúc tuần hoàn, phi tuần hoàn và gián đoạn.[10]
Các mạng mở rộng
sửaCác phức hợp axit nucleic nhỏ có thể trang bị các đầu "dính" (dễ gắn kết) và hợp lại thành những mạng tuần hoàn 2 chiều lớn hơn chứa những hình dạng lưới nhất định từ những viên gạch phân tử riêng lẻ.[10] Ví dụ sớm nhất về phương thức này sử dụng các phức hợp DX làm các viên gạch cơ bản, mỗi phức hợp DX có 4 đầu dính với trình tự khiến cho các đơn vị DX kết hợp thành các phiến phẳng 2 chiều về cơ bản có thể xem như những tinh thể 2 chiều của DNA.[14][15] Các mảng 2 chiều cũng được tạo ra từ các môtip khác nữa, bao gồm các mạng mối nối Holliday hình thoi,[16] và nhiều mảng dựa trên DX lợi dụng các ý đồ gắn kết kép.[17][18] Hai hình ảnh phía bên phải minh họa ví dụ về mạng tuần hoàn xếp gạch.
Các mảng 2 chiều có thể được thiết kế sao cho mang cấu trúc phi tuần hoàn tuân theo một thuật toán nhất định, thể hiện một dạng của điện toán DNA.[19] Các viên gạch DX có thể có trình tự đầu dính được chọn sao cho chúng đóng vai trò như những viên gạch Wang, cho phép chúng thực hiện tính toán. Người ta đã chứng minh thành công một mảng DX được tổng hợp để làm phép tính XOR; điều này cho phép DNA tạo nên những robot tế bào sinh ra một fractal được gọi là miếng đệm Sierpinski. Hình thứ 3 ở bên phải minh họa dạng mảng này.[20] Một hệ thống khác có chức năng của một mạch đếm nhị phân, biểu diễn sự tăng các số nhị phân khi nó hình thành. Các kết quả này chứng tỏ rằng tính toán có thể thực hiện đưa vào việc tổng hợp các mảng DNA.[21]
Các mảng DX cũng có thể hình thành các ống nano đường kính 4–20 nm, về cơ bản là những mạng 2 chiều uốn cong vào chính nó.[22] Các ống nano DNA này ít nhiều tương tự về kích thước và hình dạng với ống nano carbon (CNT), và trong khi chúng không có tính dẫn điện như CNT, ống nano DNA dễ chỉnh sửa và kết nối với những cấu trúc khác hơn. Một trong số nhiều phương pháp xây dựng nên ống nano DNA sử dụng một mạng các viên gạch DX uốn cong tự cuộn vào mình và khép lại thành một ống.[23] Một phương pháp khác cho phép chu vi có thể được xác định theo một cách đơn giản sử dụng những viên gạch chứa một dải, sự vững chắc của ống là một yếu tố đột hiện.[24]
Sự tạo ra các mạng 3 chiều DNA là mục tiêu từ buổi đầu của công nghệ nano DNA, nhưng lại là một trong những mục tiêu khó thực hiện nhất. Thành công trong việc sử dụng một môtip dựa trên khái niệm về tính toàn vẹn căng (tiếng Anh: tensegrity=tension, sự căng+integrity, toàn vẹn), một sự cân bằng giữa các lực căng và lực kéo, cuối cùng đã thành hiện thực vào năm 2009.[19][25]
Các cấu trúc gián đoạn
sửaCác nhà nghiên cứu đã tổng hợp được một số các phức hợp DNA 3 chiều mà mỗi phức hợp có dạng kết nối của một đa diện, chẳng hạn như các khối lập phương hoặc khối 8 mặt, nghĩa là các DNA kép đi theo các cạnh của một đa giác với các mối nối DNA ở mỗi đỉnh.[26] Những bằng chứng đầu tiên về đa giác DNA tốn rất nhiều công sức, đòi hỏi những bước thắt nối và tổng hợp pha rắn để tạo nên những đa giác nối dây.[27] Những công trình về sau tổng hợp được những đa diện dễ dàng hơn nhiều. Chúng bao gồm một hình 8 mặt DNA tạo từ một dải dài duy nhất gập lại thành cấu hình chính xác,[28] và một tứ diện có thể tạo từ 4 dải DNA trong chỉ một bước, được mô tả trên hình ở đầu bài viết này.[1]
Các cấu trúc nano có hình dạng bất kỳ, không tuần hoàn sử dụng phương pháp origami DNA. Các cấu trúc này bao gồm một dải virus tự nhiên làm "giàn đỡ", gấp thành hình dạng mong muốn bằng những dải "đinh kẹp" ngắn được thiết kế có tính toán. Phương pháp này có những ưu điểm như dễ thiết kế, bởi trình tự chuỗi được xác định trước bởi trình tự chuỗi giàn đỡ, và không đòi hỏi độ tinh khiết cao cũng như cấu tạo thành phần chính xác của dải như những phương pháp khác. Origami DNA trước tiên thành công với các cấu trúc 2 chiều, đã tạo được các hình dạng như mặt cười hay một bản đồ phác họa Tây bán cầu.[26][29] Các cấu trúc rắn 3 chiều có thể chế tạo được bằng cách sử dụng những chuỗi xoắn DNA song song thành dạng tổ ong,[30] và các cấu trúc với mặt 2 chiều có thể gấp lại thành một hình khối 3 chiều rỗng, tương tự như một hộp giấy. Chúng có thể được lập trình để mở và lộ ra hoặc giải phóng một "món hàng" phân tử dưới ảnh hưởng của một kích thích bên ngoài, khiến cho chúng có thể hữu ích trong vai trò những bộ khung phân tử.[31][32]
Tổng hợp theo khuôn mẫu
sửaCác cấu trúc axit nucleic có thể tích hợp các phân tử không phải là axit nucleic, đôi khi được gọi là dị tố (tiếng Anh: heteroelements), bao gồm protein, hạt nano kim loại, chấm lượng tử, và fullerene. Điều này cho phép xây dựng các vật liệu và thiết bị với phạm vi chức năng lớn hơn nhiều nếu chỉ dùng axit nucleic. Mục đích là dùng phép tự tổng hợp của các cấu trúc axit nucleic để làm khuôn mẫu cho việc tổng hợp các hạt nano cấy trên chúng, kiểm soát vị trí và có thể là cả định hướng của chúng.[26][33] Phần nhiều trong những ý đồ như vậy sử dụng một cơ chế gắn cộng hóa trị, bằng các oligonucleotide với nhóm chức amide hoặc thiol như những tay cầm để gắn kết các dị tố. Ý đồ liên kết cộng hóa trị này đã được sử dụng để sắp xếp các hạt nano vàng trên một mảng dựa trên DX,[34] và sắp xếp các phân tử protein streptavidin lên một mảng DX.[35] Một cơ chế cấy cộng hóa trị sử dụng các polyamide Dervan trên một mảng DX đã sắp xếp được các protein streptavidin theo một hình dạng nhất định trên một mảng DX.[36] Các ống nano carbon đã được cấy lên các mảng DNA theo một hình dạng cho phép hệ thống hoạt động như một thiết bị điện tử phân tử, một transistor hiệu ứng trường ống nano carbon.[37] Hơn nữa, có những phương pháp kim loại hóa axit nucleic, trong đó một kim loại thế chỗ axit nucleic mang hình dạng thông thường của cấu trúc axit nucleic ban đầu,[38] cùng những ý đồ sử dụng các cấu trúc nano axit nucleic làm các mặt nạ in thạch bản, chuyển hình dạng của chúng sang một bề mặt rắn.[39]
Công nghệ nano DNA động lực
sửaCông nghệ nano DNA động lực tập trung vào việc tạo ra các hệ axit nucleic với các chức năng động lực được thiết kế liên quan tới cấu trúc chung của chúng, chẳng hạn cho tính toán hoặc chuyển động cơ học. Có ít nhiều chồng lấn giữa các nhánh công nghệ nano DNA cấu trúc và động lực, bởi vì các cấu trúc có thể tạo thành thông qua ủ nhiệt và sau đó tái cấu trúc một cách động lực, hoặc có thể được chế tạo để hình thành theo con đường động lực ngay từ đầu.[26][40]
Các thiết bị cơ học nano
sửaCác phức hợp DNA có thể thay đổi hình dạng dưới những kích thích nhất định, khiến cho chúng trở thành ứng viên cho robot nano. Các cấu trúc này ban đầu được tạo theo cách giống như các cấu trúc tĩnh trong công nghệ DNA cấu trúc, những được thiết kế sao cho một sự tái cấu trúc động lực học có thể xảy ra sau sự tổng hợp ban đầu.[9][40] Thiết bị đầu tiên có khả năng như vậy sử dụng chuyển vị giữa các dạng B-DNA và Z-DNA để đáp ứng lại một thay đổi trong các điều kiện đệm nhờ trải qua một chuyển động xoắn.[41] Tuy nhiên sự phụ thuộc vào các điều kiện đệm này khiến tất cả các thiết bị thay đổi trạng thái cùng lúc, một điều không mong muốn. Các hệ thống về sau có thể thay đổi trạng thái dựa trên sự tồn tại của các dải kiểm soát, cho phép nhiều thiết bị có thể vận hành độc lập trong dung dịch. Một vài ví dụ về những hệ như vậy là một thiết kế "kẹp phân tử" có các trạng thái mở và đóng,[42] một thiết bị có thể bật từ dạng xuyên chéo sóng đôi (tiếng Anh: paranemic-crossover, ký hiệu PX) sang dạng mối nối kép (JX2), thông qua chuyển động quay trong quá trình,[43] và một mảng hai chiều có thể giãn ra và co lại tùy theo các dải kiểm soát.[44] Người ta cũng chế tạo được các cấu trúc có thể đóng hoặc mở một cách động lực, có khả năng đảm nhận vai trò như một chiếc khung để giải phóng hoặc lộ ra một phân tử chuyên chở được kích hoạt khi mở ra.[31][45][46]
DNA di chuyển là một lớp các cỗ máy nano axit nucleic thể hiện chuyển động có định hướng dọc theo một đường thẳng vạch sẵn. Đã có hàng loạt ý đồ liên quan tới chúng.[40] Một chiến lược trong số đó là kiểm soát chuyển động của thiết bị di chuyển dọc theo rãnh bằng các dải kiểm soát cần thêm vào chuỗi.[47][48] Một cách tiếp cận khác là lợi dụng các enzym giới hạn hoặc deoxyribozym để chia tách các dải và khiến thiết bị tiến về phía trước, cách này có lợi thể là có thể vận hành tự động.[49][50] Gần đây một hệ thống đã có thể di chuyển trên một bề mặt 2 chiều thay vì một rãnh thẳng, và chứng minh được khả năng nhặt có chọn lọc "hàng hóa" và di chuyển chúng.[51] Ngoài ra, một thiết bị dịch chuyển thẳng đã thể hiện khả năng tổng hợp dựa trên bản mẫu DNA trong lúc di chuyển trên đường, cho phép các tổng hợp hóa học nhiều bước có thể chỉ đạo bởi thiết bị.[52]
Bậc dịch chuyển dải
sửaCác bậc phản ứng dịch chuyển dải có thể sử dụng cho mục đích tính toán hoặc cấu trúc. Một phản ứng dịch chuyển dải riêng lẻ liên quan tới sự tiết lộ một trình tự mới đáp ứng sự hiện diện của một dải khởi phát. Nhiều phản ứng như vậy có thể liên kết lại thành một cấu trúc bậc thác với mỗi trình tự đầu ra mới được tiết lộ của một phản ứng lại khởi phát một phản ứng dịch chuyển dải ở gần đó. Điều này cho phép xây dựng các mạng lưới phản ứng hóa học với nhiều thành tố, thể hiện những khả năng tính toán và xử lý thông tin phức tạp. Các bậc này thuận về mặt nhiệt động do tạo nên các cặp base mới, và entropy của hệ tăng từ các phản ứng giải tổng hợp. Các bậc dịch chuyển chuỗi cho phép các quá trình tổng hợp hoặc tính toán vận hành trong điều kiện đẳng nhiệt, trái với đòi hỏi của tổng hợp axit nucleic truyền thống với phải nhiệt độ tăng lên sau đó từ từ giảm đi để đảm bảo sự hình thành chính xác các cấu trúc mong muốn. Chúng cũng hỗ trợ chức năng xúc tác của chuỗi khởi phát.[9][53]
Các phức hợp dịch chuyển dải cho phép các cổng logic phân tử có thể thực hiện tính toán phức tạp.[54] Không giống như máy tính điện tử truyền thống, sử dụng dòng điện làm đầu vào và đầu ra, máy tính phân tử sử dụng nồng độ của các chất hóa học nhất định làm tín hiệu. Trong trường hợp của mạch dịch chuyển dải axit nucleic, tín hiệu là sự hiện diện của các dải axit nucleic được giải phóng hoặc tiêu thụ trong các sự kiện kết hợp và giải kết hợp với các dải khác trong cấu trúc phức dịch chuyển. Cách tiếp cận này đã được sử dụng để chế tạo thành công các cổng logic như AND, OR, và NOT.[55] Gần đây, một mạch 4-bit sử dụng một hệ các cổng chứa 130 dải DNA đã chứng tỏ có thể tính toán căn bậc hai của các số nguyên từ 0-15.[56]
Cũng có thể dùng các bậc dịch chuyển dải để chế tạo các cấu trúc tổng hợp động lực học. Chúng sử dụng một cấu trúc "kẹp tóc" cho các chất phản ứng, để cho khi dải đầu vào kết nối, trình tự mới hé lộ nằm trên cùng phân tử tử thay vì giải tổng hợp. Điều này cho phép các cấu trúc kẹp tóc mới mở thêm vào một phức hợp lớn lên. Cách tiếp cận này có thể tạo nên những cấu trúc đơn giản như là mối nối ba nhánh hoặc bốn nhánh và các dendrimer.[53]
Ứng dụng
sửaCông nghệ nano DNA là một trong vài cách khả dĩ để xây dựng các cấu trúc phức tạp được thiết kế với sự kiểm soát chính xác bậc nano. Lĩnh vực này bắt đầu hé lộ ứng dụng tiềm năng trong việc giải đáp những vấn đề khoa học cơ bản trong các ngành sinh học cấu trúc và lý sinh học. Ứng dụng sớm nhất được trông đợi về mảng này, và hiện vẫn đang phát triển, liên quan tới ngành tinh thể học, với các phân tử khó tinh thể hóa trong điều kiện cô lập có thể sắp xếp vào trong những mạng axit nucleic 3 chiều, cho phép xác định cấu trúc của chúng. Một lĩnh vực khác sử dụng các thanh DNA origami để thay thế cho các tinh thể lỏng trong các thí nghiệm về ghép cặp lưỡng cực điện dư trong phổ kế NMR protein; việc sử dụng DNA có ưu thế là khác với tinh thể lỏng, chúng chịu được các chất tẩy dùng để ngưng tụ các protein màng trong dung dịch. Hơn nữa, các cấu trúc DNA origami có thể đóng góp vào các nghiên cứu lý sinh về chức năng của enzym và sự cuộn gấp protein.[10][57]
Công nghệ DNA đang hướng tới những ứng dụng tiềm năng trong thực tế. Khả năng sắp xếp các nguyên tử các của các mảng axit nucleic gợi rằng chúng có thể có ứng dụng quan trọng trong điện tử phân tử. Sự tổng hợp của một cấu trúc axit nucleic có thể dùng làm khuôn tổng hợp cho các yếu tố điện tử phân tử như các sợi dây phân tử, cung cấp một phương pháp để kiểm soát sự xếp đặt và cấu trúc tổng thể của thiết bị ở bậc nanomet như thể một bảng mạch phân tử.[26] Công nghệ nano DNA từng được ví với khái niệm về vật liệu có thể lập trình (tiếng Anh: programmable matter). do sự liên hệ giữa tính toán và tính chất vật liệu của nó.
Trong một nghiên cứu thực hiện ở Đại học Aarhus (Đan Mạch), các nhà nghiên cứu đã chế tạo thành công một "hộp" origami DNA. Hộp này đặc biệt ở chỗ có thể đóng mở nhiều lần dưới sự kích thích của một số "chìa khóa" DNA hoặc RNA. Hộp này có triển vọng dùng để vận tải phân tử (trong y học nano) hoặc kiểm soát chức năng của những phân tử riêng lẻ.[58]
Có những tiềm năng ứng dụng công nghệ DNA nano trong lĩnh vực y học nano, sử dụng khả năng thực hiện tính toán tương thích sinh học để chế tạo những "viên thuốc thông minh" cho việc vận chuyển thuốc tới đích đến là những cơ quan nhất định trong cơ thể. Một hệ thống như vậy sử dụng một hộp DNA rỗng chứa những protein có thể gây nên sự chết rụng tế bào mà chỉ mở ra khi ở gần một tế bào ung thư.[57][59] Cũng có những mối quan tâm khác trong việc đưa các cấu trúc nhân tạo này vào các tế bào vi khuẩn tổng hợp, có thể sử dụng các RNA phiên mã cho việc tổng hợp, mặc dù người ta vẫn chưa biết liệu các cấu trúc phức hợp này có thể gấp lại hoặc tổng hợp trong tế bào chất của tế bào đích hay không. Nếu thành công, điều này có thể cho phép tạo nên tiến hóa định hướng trong các cấu trúc axit nucleic.[26] Các nhà khoa học tại Đại học Oxford đã tường thuật sự tự tổng hợp của bốn dải DNA ngắn thành một dạng khung có khả năng đi vào bên trong tế bào và tồn tại trong vòng 48 giờ. Các mặt tứ diện DNA này có khả năng duy trì liên lạc trong các tế bào thận người được nuôi cấy trong phòng thí nghiệp bất chấp sự tấn công của các enzym. Thí nghiệm này cho thấy khả năng vận chuyển thuốc vào bên trong tế bào sống sử dụng các khung DNA.[60][61] Một tứ diện DNA tương tự đã được dùng để vận chuyển chất Can thiệp RNA (RNAi) vào một con chuột thí nghiệm, theo một báo cáo của một nhóm nghiên cứu từ MIT. Sự vận chuyển chất can thiệp RNA cho việc điều trị đã bước đầu thành công bằng cách sử dụng polymer hoặc lipid, nhưng vẫn có những giới hạn về độ an toàn và độ nhắm đích chính xác, cũng như tuổi thọ ngắn trong các tế bào máu.[62][63]
Thiết kế
sửaCác cấu trúc nano DNA phải được thiết kế theo ý đồ sao cho các dải axit nucleic đơn lẻ tự kết hợp thành những cấu trúc mong muốn. Quá trình này thường bắt đầu với việc xác định đặc tính của một cấu trúc đích hoặc chức năng mong muốn. Sau đó, cấu trúc bậc hai của một phức hợp đích được xác định, bao gồm thông tin về sự sắp xếp các dải axit nucleic trong cấu trúc, và những phần nào của những dải đó sẽ gắn vào với nhau. Bước cuối cùng là thiết kế cấu trúc bậc một, tức xác định trình tự chuỗi thực sự của mỗi từng axit nucleic.[22][64]
Thiết kế cấu trúc
sửaBước đầu tiên trong thiết kế một cấu trúc nano axit nucleic là xác định xem làm thế nào một cấu trúc cho trước có thể biểu diễn bằng một sự sắp xếp nhất định các dải axit nucleic. Bước thiết kế này xác định cấu trúc bậc hai, hay vị trí của các cặp base giữ cho các dải đơn lẻ gắn kết với nhau theo hình dạng mong muốn.[22] Một vài cách tiếp cận đã được chứng minh bằng thực nghiệm:
- Cấu trúc xếp gạch. Cách tiếp cận này phân nhỏ cấu trúc đích ra thành những đơn vị nhỏ hơn với liên kết mạnh giữa các dải chứa trong từng đơn vị, và các tương tác yếu giữa các đơn vị với nhau. Nó thường được dùng để tạo nên các mạng tuần hoàn, nhưng cũng có thể áp dụng cho tự tổng hợp theo thuật toán, tạo nên một cơ sở cho tính toán DNA. Đây là chiến lược thiết kế có ưu thế nổi trội dùng trong những năm từ giữa thập niên 1990 tới giữa thập niên 2000, trước khi phương pháp origami DNA xuất hiện.[22][65]
- Cấp trúc gấp. Một cách tiếp cận mới hơn, tạo nên cấu trúc nano từ một dải dài duy nhất. Dải dài này có thể có một trình tự được thiết kế để gấp lại do tương tác với chính nó, hoặc có thể gấp lại nhờ những dải ngắn hơn gọi là "ghim kẹp". Phương pháp sau (dùng "ghim kẹp") gọi là origami DNA, có thể tạo nên những hình dạng bậc nano 2 hoặc 3 chiều (xem mục Cấu trúc rời rạc).[26][29]
- Tổng hợp động lực. Cách tiếp cận này trực tiếp kiểm soát quá trình động lực học của sự tự tổng hợp DNA, xác định rõ tất cả các bước trung gian trong cơ chế phản ứng chứ không chỉ sản phẩm cuối cùng. Điều này thực hiện được nhờ sử dụng những vật liệu ban đầu có cấu trúc "kẹp tóc"; chúng tổng hợp thành hình dạng cuối cùng trong một phản ứng bậc thác, theo một trật tự nhất định(xem mục Bậc dịch chuyển dải ở trên). Cách tiếp cận này có lợi thế là xảy ra đẳng nhiệt-tức ở một nhiệt độ không đổi. Điều này trái với các cách tiếp cận nhiệt động, vốn đòi hỏi một bước ủ nhiệt mà trong đó cần thay đổi nhiệt độ để kích hoạt sự tổng hợp và khuyến khích việc hình thành chính xác các cấu trúc mong muốn.[26][53]
Thiết kế chuỗi
sửaSau khi bước thiết kế cấu trúc bậc hai cho phức hợp đích bằng một trong các cách tiếp cận ở trên hoàn thành, người ta cần phải tạo ra một trình tự thực sự cho chuỗi nucleotide. Thiết kế axit nucleic là quá trình gán một trình tự chuỗi nhất định cho một dải thành phần của cấu trúc sao cho chúng sẽ gắn kết thành hình dạng mong muốn. Hầu hết các phương pháp có mục đích thiết kế chuỗi sao cho cấu trúc đích có năng lượng nhỏ nhất, và do đó được ưu tiên về mặt nhiệt động, trong khi các cấu trúc tổng hợp sai phải có năng lượng lớn hơn và do đó không được ưu tiên. Điều này được thực hiện hoặc nhờ các phương pháp heuristic đơn giản và nhanh hơn như cực tiểu hóa đối xứng chuỗi, hoặc bằng cách sử dụng mô hình nhiệt động lân cận đầy đủ, vốn chính xác hơn nhưng chậm hơn và đòi hỏi năng lực tính toán (của máy tính) nhiều hơn. Các mô hình hình học được dùng kiểm tra cấu trúc bậc ba của các cấu trúc nano và để đảm bảo rằng các phức hợp đó không bị kéo căng quá mức.[64][66]
Thiết kế axit nucleic có mục đích tương tự như thiết kế protein. Trong cả hai quá trình, trình tự chuỗi đơn phân phải đảm bảo năng lượng cực tiểu cho cấu trúc đích. thiết kế axit nucleic có lợi thế là dễ dàng về mặt tính toán hơn protein, bởi quy tắc ghép cặp chuỗi đơn giản là đủ để tiên đoán ưu tiên năng lượng, không cần tới những thông tin chi tiết về gấp xếp 3 chiều tổng thể của cấu trúc. Điều này cho phép sử dụng các phương pháp heuristic đem lại những thiết kế tỏ ra bền vững trong thực nghiệm. Tuy nhiên, các cấu trúc axit nucelic ít hữu dụng bằng protein về mặt chức năng bởi protein có khả năng gấp thành những cấu trúc hết sức phức tạp, cũng như sự hạn chế về đa dạng hóa tính của bốn loại nucleotide so với 20 loại amino acid trong protein.[66]
Vật liệu và kỹ thuật
sửaTrình tự chuỗi của các dải DNA tạo nên cấu trúc đích được thiết kế bằng máy tính, sử dụng các phần mềm làm mẫu phân tử và làm mẫu nhiệt động.[64][66] Các phân tử được tổng hợp dựa trên các phương pháp tổng hợp oligonucleotide thông thường, thường tự động hóa trong một bộ tổng hợp oligonucleotide, và các dải với trình tự tùy biến hiện có sẵn dưới dạng sản phẩm thương mại.[67] Các chuỗi có thể được làm sạch bằng cách làm biến chất điện di gel nếu cần thiết,[68] và nồng độ có thể xác định được chính xác qua một trong vài phương pháp định tính axit nucleic sử dụng phổ kế hấp thụ tử ngoại.[69]
Các cấu trúc đích sau khi hình thành hoàn toàn sẽ được kiểm chứng bằng phương pháp điện di gel tự nhiên, cung cấp thông tin về hình dạng và kích thước của các phức hợp axit nucleic. Một phép xét nghiệm dịch chuyển độ linh động điện di có thể khẳng định liệu cấu trúc đó có bao gồm tất cả các dải mong muốn.[70] Đôi khi người ta cũng dùng các phương pháp đánh dấu huỳnh quang và truyền năng lượng cộng hưởng Förster(FRET)để xác định cấu trúc của các phức hợp.[71]
Kính hiển vi lực nguyên tử có khả năng chụp ảnh trực tiếp các cấu trúc axit nuleic, rất thích hợp với các cấu trúc hai chiều, nhưng ít hữu dụng cho các cấu trúc ba chiều rời rạc bởi tương tác giữa mũi dò của kính hiển vi với cấu trúc axit nucleic vốn dễ đứt gãy; trong trường hợp này người ta thường dùng kính hiển vi điện tử truyền qua và kính hiển vi điện tử cryo. Các mạng 3 chiều mở rộng được phân tích bằng tinh thể học tia X.[72][73]
Lịch sử
sửaNadrian Seeman đã lập nên nền tảng khái niệm cho công nghê nano DNA vào đầu những năm 1980.[74] Động lực ban đầu của Seeman là tạo ra một mạng DNA ba chiều để định hướng những phân tử lớn hơn, giúp đơn giản hóa việc nghiên cứu tinh thể học các phân tử này do loại bỏ được quá trình phức tạp cần để thu được những tinh thể tinh khiết. ý tưởng này được là đến với ông vào cuối năm 1980, sau khi ông nhận ra sự tương tự giữa bản khắc gỗ Chiều sâu của M. C. Escher và một mảng mối nối DNA sáu nhánh.[3][75] Vào lúc bấy giờ người ta đã biết đến một số cấu trúc DNA phân nhánh tự nhiên, bao gồm nhánh chĩa tái tạo DNA và mối nối Holliday di động, nhưng tầm nhìn của Seeman những mối nối axit nucleic bất động có thể tạo ra bằng cách thiết kế chính xác trình dự dải để loại bỏ đối xứng trong phân tự tổng hợp, và rằng những mối nối bất động đó có thể, về mặt nguyên lý, kết hợp thành những mạng tinh thể vững bền. Bài báo lý thuyết đầu tiên đề xuất sơ đồ này được công bố vào năm 1982, và một mối nối DNA bất động đầu tiên được chứng minh bằng thực nghiện năm sau đó.[5][26]
Năm 1991, phòng thí nghiệm của Seeman ở Đại học New York tuyên bố tổng hợp thành công một khối lập phương DNA, cấu trúc tổng hợp ba chiều đầu tiên từ DNA, công trình này đã đem lại cho ông giải Feynman về Công nghệ Nano năm 1995. Sau đó một khối bát diện DNA chặt cụt cũng xuất hiện. Tuy nhiên, cộng đồng nghiên cứu sớm nhận ra rằng những cấu trúc này, với hình dạng đa diện cùng các mối nối co giãn nằm ở đỉnh, không đủ vững chắc để tạo nên những mạng ba chiều mở rộng. Seeman phát triển motip xuyên chéo kép (DX) bền hơn, vào năm 1998, cộng tác với Erik Winfree, công bố thành công trong việc tạo ra những mạng hai chiều từ những viên gạch DX.[3][74][76] Những cấu trúc xếp gạch này có lợi thế là chúng cung cấp khả năng thực hiện tính toán DNA, như được chứng minh bởi Winfree và Paul Rothemund trong một bài báo năm 2004 về sự tự tổng hợp theo thuật toán của một cấu trúc miếng đệm Sierpinski, giúp họ nhận được giải Feymann về Công nghệ Nano năm 2009. Viễn kiến quan trọng của Winfree là các viên gạch DX có thể sử dụng làm các như các viên gạch Wang, nghĩa là sự tổng hợp chúng có khả năng thực hiện phép tính toán.[74] Sự tổng hợp một mạng ba chiều cuối cùng cũng được Seeman thực hiện năm 2009, gần 30 năm sau khi ông xác định mục tiêu đạt được nó.[57]
Những khả năng mới tiếp tục được khám phá cho các cấu trúc DNA tổng hợp trong suốt những năm 2000. Cỗ máy nano DNA đầu tiên-một môtip thay đổi cấu trúc của nó theo một đầu vào-được chứng minh năm 1999, cũng do nhóm nghiên cứu của Seeman. Một hệ thống cải tiến, thiết bị axit nucleic đầu tiên sử dụng dịch chuyển can thiệp chỗ đứng chân, được chứng minh bởi Bernard Yurke năm sau đó. Bước tiến bộ tiếp theo là chyển đổi dịch chuyển này thành chuyển động cơ học, vào năm 2004-2005, một số hệ thống DNA di chuyển đã được chứng minh bởi các nhóm Seeman, Niles Pierce, Andrew Turberfield, và Chengde Mao.[40] Ý tưởng sử dụng các mảng DNA để làm khuôn cho tổng hợp các phân tử khác như các hạt nano và protein, do Bruche Robinson và Seeman đề xuất năm 1987,[77] đã thành hiện thực năm 2006-2007 bởi các nhóm của Hao Yan, Peter Dervan, và Thomas LaBean.[5][33]
Năm 2006, Rothemund lần đầu tiên chứng minh kỹ thuật origami DNA cho phép chế tạo dễ dàng những cấu trúc DNA xếp gấp vững chãi với hình dạng tùy ý. Rothemund xem phương pháp này như trung gian về mặt khái niệm giữa các mạng DX của Seeman sử dụng nhiều dải ngắn, và bát diện DNA của William Shih bao gồm hầu như chỉ một dải rất dài. Origami của Rothemund chứa một dải dài gấp lại nhờ sự hỗ trợ của một số dải ngắn. Phương pháp này cho phép chế tạo những cấu trúc lớn hơn nhiều so với trước kia, và ít phức tạp về mặt kỹ thuật ttrong việc thiết kế và tổng hợp.[76] Origami DNA lên trang bìa của tạp chí Nature ngày 15 tháng 3 năm 2006.[29] Sau đó, vào năm 2009, Douglas đã chế tạo được origami dạng đặc 3 chiều,[30] trong khi các nhóm của Jørgen Kjems và Yan tạo được cấu trúc rỗng làm từ các mặt hai chiều.[57]
Ban đầu công nghệ nano DNA được đón nhận với ít nhiều ngờ vực do việc sử dụng axit nucleic ngoài lĩnh vực sinh học để làm vật liệu xây dựng cấu trúc hay thực hiện tính toán, cũng như ưu thế của những thí nghiệm chứng minh nguyên lý mở rộng các triển vọng của lĩnh vực nhưng vẫn còn xa với ứng dựng thực. Bài báo cột mốc năm 1991 Seeman về tổng hợp khối lập phương DNA đã bị tờ Science từ chối sau dù một người phê bình ca ngợi tính độc đáo của nó nhưng một người khác lại phê phán rằng nó thiếu liên quan tới sinh học. Tuy nhiên tới đầu thập niên 2010, lĩnh vực đã mở rộng các tiềm năng của nó tới mức mà ứng dụng cho nghiên cứu khoa học cơ bản đã được hiện thực hóa, và các ứng dụng thực tế trong y học và các ngành khác bắt đầu trở nên khả thi.[57][78] Lĩnh vực này đã lớn lên từ chỗ chỉ có vài phòng thí nghiệm tích cực nghiên cứu vào năm 2001 lên tới ít nhất 60 phòng thí nghiệm như thế vào năm 2010, tăng cường vốn trí tuệ đầu tư cũng như số lượng những tiến bộ khoa học trong thập kỉ này.[19]
Tham khảo
sửa- ^ a b DNA polyhedra: Goodman, Dmssel P.; Schaap, Iwan A. T.; Tardin, C. F.; Erben, Christof M.; Berry, Richard M.; Schmidt, C.F.; Turberfield, Andrew J. (ngày 9 tháng 12 năm 2005). “Rapid chiral assembly of rigid DNA building blocks for molecular nanofabrication”. Science. 310 (5754): 1661–1665. Bibcode:2005Sci...310.1661G. doi:10.1126/science.1120367. PMID 16339440.
- ^ a b Overview: Mao, Chengde (tháng 12 năm 2004). “The emergence of complexity: lessons from DNA”. PLoS Biology. 2 (12): 2036–2038. doi:10.1371/journal.pbio.0020431. PMC 535573. PMID 15597116.
- ^ a b c d e Overview: Seeman, Nadrian C. (tháng 6 năm 2004). “Nanotechnology and the double helix”. Scientific American. 290 (6): 64–75. doi:10.1038/scientificamerican0604-64. PMID 15195395.
- ^ Background: Pelesko, John A. (2007). Self-assembly: the science of things that put themselves together. New York: Chapman & Hall/CRC. tr. 5, 7. ISBN 978-1-58488-687-7.
- ^ a b c d e f Overview: doi:10.1146/annurev-biochem-060308-102244
Hoàn thành chú thích này - ^ Background: Long, Eric C. (1996). “Fundamentals of nucleic acids”. Trong Hecht, Sidney M (biên tập). Bioorganic chemistry: nucleic acids. New York: Oxford University Press. tr. 4–10. ISBN 0-19-508467-5.
- ^ RNA nanotechnology: Chworos, Arkadiusz; Severcan, Isil; Koyfman, Alexey Y.; Weinkam, Patrick; Oroudjev, Emin; Hansma, Helen G.; Jaeger, Luc (2004). “Building Programmable Jigsaw Puzzles with RNA”. Science. 306 (5704): 2068–2072. Bibcode:2004Sci...306.2068C. doi:10.1126/science.1104686. PMID 15604402.Quản lý CS1: nhiều tên: danh sách tác giả (liên kết)
- ^ RNA nanotechnology: Guo, Peixuan (2010). “The Emerging Field of RNA Nanotechnology”. Nature Nanotechnology. 5 (12): 833–842. Bibcode:2010NatNa...5..833G. doi:10.1038/nnano.2010.231. PMC 3149862. PMID 21102465.
- ^ a b c d Dynamic DNA nanotechnology: doi:10.1038/nchem.957
Hoàn thành chú thích này - ^ a b c d Structural DNA nanotechnology: Seeman, Nadrian C. (tháng 11 năm 2007). “An overview of structural DNA nanotechnology”. Molecular Biotechnology. 37 (3): 246–257. doi:10.1007/s12033-007-0059-4. PMC 3479651. PMID 17952671.
- ^ Dynamic DNA nanotechnology: doi:10.1016/j.copbio.2006.10.004
Hoàn thành chú thích này - ^ Other arrays: Strong, Michael (tháng 3 năm 2004). “Protein Nanomachines”. PLoS Biology. 2 (3): e73. doi:10.1371/journal.pbio.0020073. PMC 368168. PMID 15024422.
- ^ doi:10.1126/science.1089389
Hoàn thành chú thích này - ^ DX arrays: Winfree, Erik; Liu, Furong; Wenzler, Lisa A.; Seeman, Nadrian C. (ngày 6 tháng 8 năm 1998). “Design and self-assembly of two-dimensional DNA crystals”. Nature. 394 (6693): 529–544. Bibcode:1998Natur.394..539W. doi:10.1038/28998. PMID 9707114.
- ^ DX arrays: Liu, Furong; Sha, Ruojie; Seeman, Nadrian C. (ngày 10 tháng 2 năm 1999). “Modifying the surface features of two-dimensional DNA crystals”. Journal of the American Chemical Society. 121 (5): 917–922. doi:10.1021/ja982824a.
- ^ Other arrays: Mao, Chengde; Sun, Weiqiong; Seeman, Nadrian C. (ngày 16 tháng 6 năm 1999). “Designed two-dimensional DNA Holliday junction arrays visualized by atomic force microscopy”. Journal of the American Chemical Society. 121 (23): 5437–5443. doi:10.1021/ja9900398.
- ^ Other arrays: Constantinou, Pamela E.; Wang, Tong; Kopatsch, Jens; Israel, Lisa B.; Zhang, Xiaoping; Ding, Baoquan; Sherman, William B.; Wang, Xing; Zheng, Jianping; Sha, Ruojie; Seeman, Nadrian C. (ngày 21 tháng 9 năm 2006). “Double cohesion in structural DNA nanotechnology”. Organic and Biomolecular Chemistry. 4 (18): 3414–3419. doi:10.1039/b605212f. PMC 3491902. PMID 17036134.Quản lý CS1: sử dụng tham số tác giả (liên kết)
- ^ Other arrays: Mathieu, Frederick; Liao, Shiping; Kopatsch, Jens; Wang, Tong; Mao, Chengde; Seeman, Nadrian C. (tháng 4 năm 2005). “Six-helix bundles designed from DNA”. Nano Letters. 5 (4): 661–665. Bibcode:2005NanoL...5..661M. doi:10.1021/nl050084f. PMC 3464188. PMID 15826105.
- ^ a b c History: Seeman, Nadrian (ngày 9 tháng 6 năm 2010). “Structural DNA nanotechnology: growing along with Nano Letters”. Nano Letters. 10 (6): 1971–1978. Bibcode:2010NanoL..10.1971S. doi:10.1021/nl101262u. PMC 2901229. PMID 20486672.
- ^ Algorithmic self-assembly: Rothemund, Paul W. K.; Papadakis, Nick; Winfree, Erik (tháng 12 năm 2004). “Algorithmic self-assembly of DNA Sierpinski triangles”. PLoS Biology. 2 (12): 2041–2053. doi:10.1371/journal.pbio.0020424. PMC 534809. PMID 15583715.
- ^ Algorithmic self-assembly: Barish, Robert D.; Rothemund, Paul W. K.; Winfree, Erik (tháng 12 năm 2005). “Two computational primitives for algorithmic self-assembly: copying and counting”. Nano Letters. 5 (12): 2586–2592. Bibcode:2005NanoL...5.2586B. doi:10.1021/nl052038l. PMID 16351220.
- ^ a b c d Design: doi:10.1002/anie.200502358
Hoàn thành chú thích này - ^ DNA nanotubes: Rothemund, Paul W. K.; Ekani-Nkodo, Axel; Papadakis, Nick; Kumar, Ashish; Fygenson, Deborah Kuchnir & Winfree, Erik (ngày 22 tháng 12 năm 2004). “Design and Characterization of Programmable DNA Nanotubes”. Journal of the American Chemical Society. 126 (50): 16344–16352. doi:10.1021/ja044319l. PMID 15600335.Quản lý CS1: sử dụng tham số tác giả (liên kết)
- ^ DNA nanotubes: Yin, P.; Hariadi, R. F.; Sahu, S.; Choi, H. M. T.; Park, S. H.; Labean, T. H.; Reif, J. H. (ngày 8 tháng 8 năm 2008). “Programming DNA Tube Circumferences”. Science. 321 (5890): 824–826. Bibcode:2008Sci...321..824Y. doi:10.1126/science.1157312. PMID 18687961.
- ^ Three-dimensional arrays: Zheng, Jianping; Birktoft, Jens J.; Chen, Yi; Wang, Tong; Sha, Ruojie; Constantinou, Pamela E.; Ginell, Stephan L.; Mao, Chengde; Seeman, Nadrian C. (ngày 3 tháng 9 năm 2009). “From molecular to macroscopic via the rational design of a self-assembled 3D DNA crystal”. Nature. 461 (7260): 74–77. Bibcode:2009Natur.461...74Z. doi:10.1038/nature08274. PMC 2764300. PMID 19727196.
- ^ a b c d e f g h i Overview: doi:10.1038/nnano.2011.187
Hoàn thành chú thích này - ^ DNA polyhedra: Zhang, Yuwen; Seeman, Nadrian C. (ngày 1 tháng 3 năm 1994). “Construction of a DNA-truncated octahedron”. Journal of the American Chemical Society. 116 (5): 1661–1669. doi:10.1021/ja00084a006.
- ^ DNA polyhedra: Shih, William M.; Quispe, Joel D.; Joyce, Gerald F. (ngày 12 tháng 2 năm 2004). “A 1.7-kilobase single-stranded DNA that folds into a nanoscale octahedron”. Nature. 427 (6975): 618–621. Bibcode:2004Natur.427..618S. doi:10.1038/nature02307. PMID 14961116.
- ^ a b c DNA origami: Rothemund, Paul W. K. (ngày 16 tháng 3 năm 2006). “Folding DNA to create nanoscale shapes and patterns”. Nature. 440 (7082): 297–302. Bibcode:2006Natur.440..297R. doi:10.1038/nature04586. PMID 16541064.
- ^ a b DNA origami: Douglas, Shawn M.; Dietz, Hendrik; Liedl, Tim; Högberg, Björn; Graf, Franziska; Shih, William M. (ngày 21 tháng 5 năm 2009). “Self-assembly of DNA into nanoscale three-dimensional shapes”. Nature. 459 (7245): 414–418. Bibcode:2009Natur.459..414D. doi:10.1038/nature08016. PMC 2688462. PMID 19458720.
- ^ a b DNA boxes: Andersen, Ebbe S.; Dong, Mingdong; Nielsen, Morten M.; Jahn, Kasper; Subramani, Ramesh; Mamdouh, Wael; Golas, Monika M.; Sander, Bjoern; và đồng nghiệp (ngày 7 tháng 5 năm 2009). “Self-assembly of a nanoscale DNA box with a controllable lid”. Nature. 459 (7243): 73–76. Bibcode:2009Natur.459...73A. doi:10.1038/nature07971. PMID 19424153.
- ^ DNA boxes: Ke, Yonggang; Sharma, Jaswinder; Liu, Minghui; Jahn, Kasper; Liu, Yan; Yan, Hao (ngày 10 tháng 6 năm 2009). “Scaffolded DNA origami of a DNA tetrahedron molecular container”. Nano Letters. 9 (6): 2445–2447. Bibcode:2009NanoL...9.2445K. doi:10.1021/nl901165f. PMID 19419184.
- ^ a b Overview: doi:10.1002/cbic.200900286
Hoàn thành chú thích này - ^ Nanoarchitecture: Zheng, Jiwen; Constantinou, Pamela E.; Micheel, Christine; Alivisatos, A. Paul; Kiehl, Richard A.; Seeman Nadrian C. (tháng 7 năm 2006). “2D Nanoparticle Arrays Show the Organizational Power of Robust DNA Motifs”. Nano Letters. 6 (7): 1502–1504. Bibcode:2006NanoL...6.1502Z. doi:10.1021/nl060994c. PMC 3465979. PMID 16834438.
- ^ Nanoarchitecture: Park, Sung Ha; Pistol, Constantin; Ahn, Sang Jung; Reif, John H.; Lebeck, Alvin R.; Dwyer, Chris; LaBean, Thomas H. (tháng 10 năm 2006). “Finite-size, fully addressable DNA tile lattices formed by hierarchical assembly procedures”. Angewandte Chemie. 118 (40): 749–753. doi:10.1002/ange.200690141.Quản lý CS1: sử dụng tham số tác giả (liên kết)
- ^ Nanoarchitecture: Cohen, Justin D.; Sadowski, John P.; Dervan, Peter B. (ngày 22 tháng 10 năm 2007). “Addressing single molecules on DNA nanostructures”. Angewandte Chemie International Edition. 46 (42): 7956–7959. doi:10.1002/anie.200702767. PMID 17763481.
- ^ Nanoarchitecture: Maune, Hareem T.; Han, Si-Ping; Barish, Robert D.; Bockrath, Marc; Goddard III, William A.; Rothemund, Paul W. K.; Winfree, Erik (tháng 1 năm 2009). “Self-assembly of carbon nanotubes into two-dimensional geometries using DNA origami templates”. Nature Nanotechnology. 5 (1): 61–66. Bibcode:2010NatNa...5...61M. doi:10.1038/nnano.2009.311. PMID 19898497.
- ^ Nanoarchitecture: doi:10.1021/nn1035075
Hoàn thành chú thích này - ^ Nanoarchitecture: doi:10.1002/anie.200460257
Hoàn thành chú thích này - ^ a b c d DNA machines: Bath, Jonathan; Turberfield, Andrew J. (tháng 5 năm 2007). “DNA nanomachines”. Nature Nanotechnology. 2 (5): 275–284. Bibcode:2007NatNa...2..275B. doi:10.1038/nnano.2007.104. PMID 18654284.
- ^ DNA machines: Mao, Chengde; Sun, Weiqiong; Shen, Zhiyong; Seeman, Nadrian C. (ngày 14 tháng 1 năm 1999). “A DNA nanomechanical device based on the B-Z transition”. Nature. 397 (6715): 144–146. Bibcode:1999Natur.397..144M. doi:10.1038/16437. PMID 9923675.
- ^ DNA machines: Yurke, Bernard; Turberfield, Andrew J.; Mills, Allen P., Jr; Simmel, Friedrich C.; Neumann, Jennifer L. (ngày 10 tháng 8 năm 2000). “A DNA-fuelled molecular machine made of DNA”. Nature. 406 (6796): 605–609. Bibcode:2000Natur.406..605Y. doi:10.1038/35020524. PMID 10949296.Quản lý CS1: sử dụng tham số tác giả (liên kết)
- ^ DNA machines: Yan, Hao; Zhang, Xiaoping; Shen, Zhiyong; Seeman, Nadrian C. (ngày 3 tháng 1 năm 2002). “A robust DNA mechanical device controlled by hybridization topology”. Nature. 415 (6867): 62–65. Bibcode:2002Natur.415...62Y. doi:10.1038/415062a. PMID 11780115.
- ^ DNA machines: doi:10.1002/ange.200351818
Hoàn thành chú thích này - ^ DNA machines: doi:10.1038/nnano.2008.3
Hoàn thành chú thích này - ^ Applications: doi:10.1126/science.1214081
Hoàn thành chú thích này - ^ DNA walkers: Shin, Jong-Shik; Pierce, Niles A. (ngày 8 tháng 9 năm 2004). “A synthetic DNA walker for molecular transport”. Journal of the American Chemical Society. 126 (35): 10834–10835. doi:10.1021/ja047543j. PMID 15339155.
- ^ DNA walkers: Sherman, William B.; Seeman, Nadrian C. (tháng 7 năm 2004). “A precisely controlled DNA biped walking device”. Nano Letters. 4 (7): 1203–1207. Bibcode:2004NanoL...4.1203S. doi:10.1021/nl049527q.
- ^ DNA walkers: Tian, Ye; He, Yu; Chen, Yi; Yin, Peng; Mao, Chengde (ngày 11 tháng 7 năm 2005). “A DNAzyme that walks processively and autonomously along a one-dimensional track”. Angewandte Chemie. 117 (28): 4429–4432. doi:10.1002/ange.200500703.
- ^ DNA walkers: Bath, Jonathan; Green, Simon J.; Turberfield, Andrew J. (ngày 11 tháng 7 năm 2005). “A free-running DNA motor powered by a nicking enzyme”. Angewandte Chemie International Edition. 44 (28): 4358–4361. doi:10.1002/anie.200501262.
- ^ Functional DNA walkers: Lund, Kyle; Manzo, Anthony J.; Dabby, Nadine; Michelotti, Nicole; Johnson-Buck, Alexander; Nangreave, Jeanette; Taylor, Steven; Pei, Renjun; Stojanovic, Milan N.; Walter, Nils G.; Winfree, Erik; Yan, Hao (ngày 13 tháng 5 năm 2010). “Molecular robots guided by prescriptive landscapes”. Nature. 465 (7295): 206–210. Bibcode:2010Natur.465..206L. doi:10.1038/nature09012. PMC 2907518. PMID 20463735.
- ^ Functional DNA walkers: He, Yu; Liu, David R. (tháng 11 năm 2010). “Autonomous multistep organic synthesis in a single isothermal solution mediated by a DNA walker”. Nature Nanotechnology. 5 (11): 778–782. Bibcode:2010NatNa...5..778H. doi:10.1038/nnano.2010.190. PMC 2974042. PMID 20935654.
- ^ a b c Kinetic assembly: Yin, Peng; Choi, Harry M. T.; Calvert, Colby R.; Pierce, Niles A. (ngày 17 tháng 1 năm 2008). “Programming biomolecular self-assembly pathways”. Nature. 451 (7176): 318–322. Bibcode:2008Natur.451..318Y. doi:10.1038/nature06451. PMID 18202654.
- ^ Fuzzy and Boolean logic gates based on DNA: doi:10.1002/smll.201402755
Hoàn thành chú thích này - ^ Strand displacement cascades: doi:10.1126/science.1132493
Hoàn thành chú thích này - ^ Strand displacement cascades: doi:10.1126/science.1200520
Hoàn thành chú thích này - ^ a b c d e History/applications: Service, Robert F. (ngày 3 tháng 6 năm 2011). “DNA nanotechnology grows up”. Science. 332 (6034): 1140–1143. doi:10.1126/science.332.6034.1140.
- ^ M. Zadegan, Reza; et, al. (2012). “Construction of a 4 Zeptoliters Switchable 3D DNA Box Origami”. ACS Nano. 6 (11): 10050–10053. doi:10.1021/nn303767b. Chú thích có tham số trống không rõ:
|1=(trợ giúp) - ^ Applications: Jungmann, Ralf; Renner, Stephan; Simmel, Friedrich C. (tháng 3 năm 2008). “From DNA nanotechnology to synthetic biology”. HFSP journal. 2 (2): 99–109. doi:10.2976/1.2896331. PMC 2645571. PMID 19404476.
- ^ Lovy, Howard (ngày 5 tháng 7 năm 2011). “DNA cages can unleash meds inside cells”. fiercedrugdelivery.com. Truy cập ngày 22 tháng 9 năm 2013.[liên kết hỏng]
- ^ Walsh, Anthony; Yin, Hai; Erben, Christoph; Wood, Matthew; Turberfield, Andrew (2011). “DNA Cage Delivery to Mammalian Cells”. ACS Nano. ACS Publications. 5 (7): 5427–5432. doi:10.1021/nn2005574. PMID 21696187.
- ^ Trafton, Anne (ngày 4 tháng 6 năm 2012). “Researchers achieve RNA interference, in a lighter package”. MIT News. Truy cập ngày 22 tháng 9 năm 2013.
- ^ Lee, Hyukjin; Lytton-Jean, Abigail; Chen, Yi; Love, Kevin; Park, Angela; Karagiannis, Emmanouil; Sehgal, Alfica; Querbes, William; và đồng nghiệp (2012). “Molecularly self-assembled nucleic acid nanoparticles for targeted in vivo siRNA delivery” (PDF). Nature Nanotechnology. Nature. 7 (6): 389–393. doi:10.1038/NNANO.2012.73. Pdf
- ^ a b c Design: Brenneman, Arwen; Condon, Anne (ngày 25 tháng 9 năm 2002). “Strand design for biomolecular computation”. Theoretical Computer Science. 287: 39–58. doi:10.1016/S0304-3975(02)00135-4.
- ^ Overview: Lin, Chenxiang; Liu, Yan; Rinker, Sherri; Yan, Hao (ngày 11 tháng 8 năm 2006). “DNA tile based self-assembly: building complex nanoarchitectures”. ChemPhysChem. 7 (8): 1641–1647. doi:10.1002/cphc.200600260. PMID 16832805.
- ^ a b c Design: Dirks, Robert M.; Lin, Milo; Winfree, Erik; Pierce, Niles A. (ngày 15 tháng 2 năm 2004). “Paradigms for computational nucleic acid design”. Nucleic Acids Research. 32 (4): 1392–1403. doi:10.1093/nar/gkh291. PMC 390280. PMID 14990744.
- ^ Methods: doi:10.1002/0471142727.mb0211s42
Hoàn thành chú thích này - ^ Methods: doi:10.1002/0471142727.mb0212s42
Hoàn thành chú thích này - ^ Methods: doi:10.1002/9780470089941.et0202s5
Hoàn thành chú thích này - ^ Methods: doi:10.1002/0471142727.mb0207s47
Hoàn thành chú thích này - ^ Methods: doi:10.1002/0471142700.nc1110s11
Hoàn thành chú thích này - ^ Methods: doi:10.1007/978-1-61779-142-0_1
Hoàn thành chú thích này - ^ Methods: Bloomfield, Victor A.; Crothers, Donald M.; Tinoco, Jr., Ignacio (2000). Nucleic acids: structures, properties, and functions. Sausalito, Calif: University Science Books. tr. 84–86, 396–407. ISBN 0-935702-49-0.Quản lý CS1: nhiều tên: danh sách tác giả (liên kết)
- ^ a b c History: Pelesko, John A. (2007). Self-assembly: the science of things that put themselves together. New York: Chapman & Hall/CRC. tr. 201, 242, 259. ISBN 978-1-58488-687-7.
- ^ History: See “Current crystallization protocol”. Nadrian Seeman Lab. Bản gốc lưu trữ ngày 30 tháng 11 năm 2012. Truy cập ngày 8 tháng 8 năm 2015.
- ^ a b DNA origami: Rothemund, Paul W. K. (2006). “Scaffolded DNA origami: from generalized multicrossovers to polygonal networks”. Trong Chen, Junghuei; Jonoska, Natasha; Rozenberg, Grzegorz (biên tập). Nanotechnology: science and computation. Natural Computing Series. New York: Springer. tr. 3–21. doi:10.1007/3-540-30296-4_1. ISBN 978-3-540-30295-7.
- ^ Nanoarchitecture: Robinson, Bruche H.; Seeman, Nadrian C. (tháng 8 năm 1987). “The design of a biochip: a self-assembling molecular-scale memory device”. Protein Engineering. 1 (4): 295–300. doi:10.1093/protein/1.4.295. PMID 3508280.
- ^ History: Hopkin, Karen (tháng 8 năm 2011). “Profile: 3-D seer”. The Scientist. Truy cập ngày 8 tháng 8 năm 2011.
Đọc thêm
sửaĐại cương:
- Seeman, Nadrian C. (tháng 6 năm 2004). “Nanotechnology and the double helix”. Scientific American. 290 (6): 64–75. doi:10.1038/scientificamerican0604-64. PMID 15195395.—Một bài viết dành cho giới không chuyên của người sáng lập nên lĩnh vực mới này
- Seeman, Nadrian C. (ngày 9 tháng 6 năm 2010). “Structural DNA nanotechnology: growing along with Nano Letters”. Nano Letters. 10 (6): 1971–1978. Bibcode:2010NanoL..10.1971S. doi:10.1021/nl101262u. PMC 2901229. PMID 20486672.—Một bài phê bình các kết quả nghiên cứu trong giai đoạn 2001–2010
- Seeman, Nadrian C. (2010). “Nanomaterials based on DNA”. Annual Review of Biochemistry. 79: 65–87. doi:10.1146/annurev-biochem-060308-102244. PMC 3454582. PMID 20222824.—Một bài phê bình toàn diện hơn bao gồm các những kết quả cũ và mới về lĩnh vực
- Service, Robert F. (ngày 3 tháng 6 năm 2011). “DNA nanotechnology grows up”. Science. 332 (6034): 1140–1143. doi:10.1126/science.332.6034.1140. and doi:10.1126/science.332.6034.1142.—Một bài báo nghiên cứu tập trung vào lịch sử của ngành và sự phát triển những ứng dụng mới
- Zadegan, Reza M.; Norton, Michael L. (tháng 6 năm 2012). “Structural DNA Nanotechnology: From Design to Applications”. Int. J. Mol. Sci. 13 (6): 7149–7162. doi:10.3390/ijms13067149. PMC 3397516. PMID 22837684.-Một bài phê bình toàn diện gần đây về lĩnh vực này
Về những đề tài cụ thể:
- Bath, Jonathan; Turberfield, Andrew J. (ngày 5 tháng 5 năm 2007). “DNA nanomachines”. Nature Nanotechnology. 2 (5): 275–284. Bibcode:2007NatNa...2..275B. doi:10.1038/nnano.2007.104. PMID 18654284.—Một bài phê bình về các thiết bị cơ học nano axit nucleic
- Feldkamp, Udo; Niemeyer, Christof M. (ngày 13 tháng 3 năm 2006). “Rational design of DNA nanoarchitectures”. Angewandte Chemie International Edition. 45 (12): 1856–76. doi:10.1002/anie.200502358. PMID 16470892.—Một bài phê bình từ quan điểm về thiết kế cấu trúc thứ cấp
- Lin, Chenxiang; Liu, Yan; Rinker, Sherri; Yan, Hao (ngày 11 tháng 8 năm 2006). “DNA tile based self-assembly: building complex nanoarchitectures”. ChemPhysChem. 7 (8): 1641–1647. doi:10.1002/cphc.200600260. PMID 16832805.—Một bài phê bình ngắn tập trung vào tổng hợp xếp gạch
- Zhang, David Yu; Seelig, Georg (tháng 2 năm 2011). “Dynamic DNA nanotechnology using strand-displacement reactions”. Nature Chemistry. 3 (2): 103–113. Bibcode:2011NatCh...3..103Z. doi:10.1038/nchem.957. PMID 21258382.—Một bài phê bình về các hệ thống DNA sử dụng cơ chế dịch chuyển dải
- Cấu trúc nano tự sao chép được làm từ DNA Lưu trữ 2017-04-18 tại Wayback Machine N.L.H (theo Phys.org, 28/5/2015). Cục Thông tin Khoa học và Công nghệ Quốc gia. Cập nhật lúc: 05 Tháng Sáu 2015 2:35:12 CH
Liên kết ngoài
sửa| Wikimedia Commons có thêm hình ảnh và phương tiện truyền tải về Công nghệ nano DNA. |
- Hiệp hội Quốc tế về Khoa học, Tính toán và Kỹ thuật Nano - International Society of Nanoscale Science, Computation and Engineering (ISNSCE)
- What is Bionanotechnology? trên YouTube - Công nghệ sinh học nano là gì? Shawn Douglas. Đã tải lên vào 07-03-2011
- Vật liệu nano tại Từ điển bách khoa Việt Nam
- Nanotechnology tại Encyclopædia Britannica (tiếng Anh)