Danh sách bộ gen lạp thể đã giải trình tự
Bộ gen lạp thể, hệ gen lạp thể hay plastome là bộ gen của lạp thể, nhóm bào quan hiện diện trong thực vật và đa dạng chủng loại nguyên sinh vật. Số lượng trình tự bộ gen lạp thể được giải mã tăng nhanh từ thập kỷ đầu tiên của thế kỷ 21. Ví dụ, có 25 bộ gen lục lạp đã được giải trình tự trong một nghiên cứu về phát sinh chủng loại phân tử.[1]

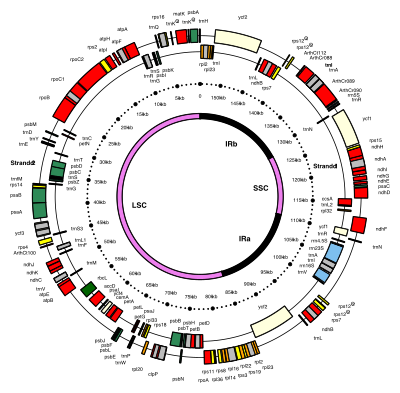

Thực vật có hoa đặc biệt xuất hiện nhiều ở những bộ gen lục lạp được giải mã. Tháng 1 năm 2017, tất cả những bộ của chúng đều đã giải mã plastome, chỉ trừ Commelinales (Bộ Thài lài), Picramniales, Huerteales, Escalloniales (Bộ Gạc nai), Bruniales, và Paracryphiales.
Một bộ sưu tập dữ liệu tất cả bộ gen lạp thể đã giải mã hoàn chỉnh đang được NCBI duy trì trong một kho tư liệu công cộng.[2]
Thực vật sửa
Rêu (theo nghĩa rộng) sửa
| Loài | Thứ | Cặp base | Gen | Chú thích | Ghi chú |
|---|---|---|---|---|---|
| Aneura mirabilis | 108.007 | [3][4] | rêu tản ký sinh; plastome chứa nhiều gen giả | ||
| Anthoceros formosae | 161.162 | 122 | [5] | rêu sừng; plastome bị RNA chỉnh sửa mạnh | |
| Marchantia polymorpha | 121.024 | [6] | rêu tản | ||
| Nothoceros aenigmaticus | 153.208 | 124 | [7] | rêu sừng | |
| Pellia endiviifolia | 120.546 | 123 | [8] | rêu tản | |
| Physcomitrella patens | 122.890 | 118 | [9] | rêu | |
| Ptilidium pulcherrimum | 119.007 | 122 | [10] | rêu tản | |
| Tortula ruralis | 122.630 | [11] | rêu |
Dương xỉ và Thạch tùng sửa
| Loài | Thứ | Cặp base | Gen | Chú thích | Họ | Ghi chú |
|---|---|---|---|---|---|---|
| Adiantum capillus-veneris | 150.568 | [12] | Pteridaceae | |||
| Alsophila spinulosa | 156.661 | 117 | [13] | Cyatheaceae | ||
| Angiopteris evecta | 153.901 | [14] | Marattiaceae | |||
| Equisetum arvense | 133.309 | Equisetaceae | ||||
| Huperzia lucidula | 154.373 | [15] | Lycopodiaceae | |||
| Isoetes flaccida | 145.303 | Isoetaceae | ||||
| Psilotum nudum | 138.829 | 117 | [16] | Psilotaceae | ||
| Selaginella uncinata | 138.829 | [17] | Selaginellaceae |
| Loài | Thứ | Cặp base | Gen | Chú thích | Họ | Ghi chú |
|---|---|---|---|---|---|---|
| Equisetum hyemale | Equisetaceae | |||||
| Lygodium japonicum | Lygodiaceae | |||||
| Marsilea crenata | Marsileaceae | |||||
| Ophioglossum californicum | Ophioglossaceae | |||||
| Selaginella moellendorffii | Selaginellaceae |
Thực vật hạt trần sửa
| Loài | Thứ | Cặp base | Gen | Chú thích | Họ | Ghi chú |
|---|---|---|---|---|---|---|
| Cryptomeria japonica | 131.810 | 114 | [18] | Cupressaceae | ||
| Cycas micronesica | [19] | Cycadaceae | ||||
| Cycas taitungensis | 163.403 | 133 | [20] | Cycadaceae | ||
| Ephedra equisetina | Ephedraceae | |||||
| Ginkgo biloba | 156.945 | 134 | [21] | Ginkgoaceae | ||
| Gnetum parvifolium | Gnetaceae | |||||
| Pinus koraiensis | 116.866 | Pinaceae | ||||
| Pinus thunbergii | 119.707 | [22] | Pinaceae | |||
| Podocarpus macrophyllus | Podocarpaceae | |||||
| Welwitschia mirabilis | 119.726 | 101 | [23] | Welwitschiaceae |
Thực vật có hoa sửa
Bảng phân loại dưới đây dự kiến sẽ sưu tập những bộ gen lạp thể hoàn chỉnh đại diện cho một phạm vi rộng nhất về kích cỡ, số lượng gen và các họ thực vật có hoa.
Tảo lục sửa
| Loài | Thứ | Cặp base | Gen | Chú thích |
|---|---|---|---|---|
| Bryopsis plumosa | 106.859 | 115 | [96] | |
| Chaetosphaeridium globosum | 131.183 | 124 | [97] | |
| Chara vulgaris | ||||
| Chlamydomonas reinhardtii | 203.395 | 99 | ||
| Chlorella vulgaris | 150.613 | 209 | [98] | |
| Chlorokybus atmophyticus | 201.763 | 70 | [99] | |
| Dunaliella salina | CCAP 19/18 | 269.044 | 102 | [100] |
| Emiliania huxleyi | 105.309 | 150 | ||
| Helicosporidium | 37.454 | 54 | [101] | |
| Leptosira terrestris | 195.081 | 117 | [102] | |
| Mesostigma viride | 42.424 | |||
| Monomastix | 114.528 | 94 | [103] | |
| Nephroselmis olivacea | 200.799 | 127 | [104] | |
| Oedogonium cardiacum | 196.547 | 103 | [105] | |
| Oltmannsiellopsis viridis | 151.933 | 105 | [106] | |
| Ostreococcus tauri | 71.666 | 86 | [107] | |
| Pseudendoclonium akinetum | 195.867 | 105 | [108] | |
| Pycnococcus provasolii | 80.211 | 98 | [103] | |
| Pyramimonas parkeae | 101.605 | 110 | [103] | |
| Scenedesmus obliquus | 161.452 | 96 | [109] | |
| Staurastrum punctulatum | [110] | |||
| Stigeoclonium helveticum | 223.902 | 97 | [111] | |
| Tydemania expeditionis | 105.200 | 125 | [96] | |
| Ulva sp. | UNA00071828 | 99.983 | 102 | [112] |
| Volvox carteri | 420.650 | 91 | [113] | |
| Zygnema circumcarinatum |
Tảo đỏ sửa
| Loài | Thứ | Cặp base | Gen | Chú thích | Ghi chú |
|---|---|---|---|---|---|
| Cyanidioschyzon merolae | 149.987 | 243 | [114] | ||
| Cyanidium caldarium | RK1 | [115] | |||
| Gracilaria tenuistipitata | var. liui | 183.883 | 238 | [116] | |
| Porphyra purpurea | |||||
| Porphyra yezoensis |
Tảo lục lam sửa
| Loài | Thứ | Cặp base | Gen | Chú thích |
|---|---|---|---|---|
| Cyanophora paradoxa |
Siêu tảo và Apicomplexa sửa
Siêu tảo là những sinh vật mang những bào quan quang hợp nguồn gốc từ sự kiện nội cộng sinh bậc hai hoặc bậc ba, cùng dạng gần không quang hợp, tựa lạp thể, hay thân thích. Apicomplexa là một ngành gồm những loài ký sinh đơn bào không quang hợp thuộc siêu nhóm nhân thực Chromalveolata, mang một bào quan lạp thể cấp hai thoái hóa.
Chromalveolata quang hợp sửa
Bộ gen lạp thể Dinoflagellata không được tổ chức thành một phân tử DNA vòng đơn giống như những bộ gen lạp thể khác, mà lại trở thành một dàn gồm những vòng nhỏ.
| Loài | Thứ | Cặp base | Gen | Chú thích | Ghi chú |
|---|---|---|---|---|---|
| Chromera velia | |||||
| Guillardia theta | 121.524 | 167 | [117] | ||
| Heterosigma akashiwo | |||||
| Odontella sinensis | 119,7 kb | 175 | |||
| Phaeodactylum tricornutum | |||||
| Thalassiosira pseudonana | 129 kb | [118] | |||
| Rhodomonas salina |
Chlorarachniophyta sửa
| Loài | Thứ | Cặp base | Gen | Chú thích |
|---|---|---|---|---|
| Bigelowiella natans | 69.166 | 87 | [119] |
Euglenophyceae sửa
| Loài | Thứ | Cặp base | Gen | Chú thích |
|---|---|---|---|---|
| Astasia longa | 73,2 kb | 84 | ||
| Euglena gracilis | 143,2 kb | 128 | [120] |
Apicomplexa sửa
| Loài | Thứ | Cặp base | Gen | Chú thích |
|---|---|---|---|---|
| Eimeria tenella | Penn State | 34,8 kb | 65 | [121] |
| Plasmodium falciparum | 34,7 kb | 68 | ||
| Theileria parva | Mugaga | 39,6 kb | 71 | |
| Toxoplasma gondii | RH | 35,0 kb | 65 |
Bộ gen nucleomorph sửa
Ở một vài sinh vật quang hợp khả năng bị tập nhiễm khi nội cộng sinh với tảo lục (Chlorophyta) hoặc tảo đỏ (Rhodophyta) đơn bào. Trong một số trường hợp này, không chỉ lục lạp từ tế bào tảo đơn bào trước đó giữ lại bộ gen của nó, mà còn kèm theo những cấu trúc tàn tích của tảo. Khi tàn dư đó bao gồm một nhân tế bào và một hệ gen nhân thì được gọi với thuật ngữ nucleomorph.
| Loài | Thứ | Cặp base | Gen | Chú thích |
|---|---|---|---|---|
| Bigelowiella natans | ||||
| Cryptomonas paramecium | ||||
| Guillardia theta | 551.264 | |||
| Hemiselmis andersenii |
Bộ gen cyanelle sửa
Loài nhân thực đơn bào Paulinella chromatophora sở hữu một bào quan (cyanelle), đại diện cho một trường hợp độc lập về việc chiếm hữu khả năng quang hợp từ sự kiện nội cộng sinh vi khuẩn lam. (Ghi chú: thuật ngữ cyanelle cũng dùng để chỉ lạp thể tảo lục lam.)
| Loài | Thứ | Cặp base | Gen | Chú thích |
|---|---|---|---|---|
| Paulinella chromatophora | 1,02 Mb | 867 | [122] |
Xem thêm sửa
Chú thích sửa
- ^ a b c d e f g h i j k l m n o p q r s t u v w x Moore MJ, Soltis PS, Bell CD, Burleigh JG, Soltis DE (2010). “Phylogenetic analysis of 83 plastid genes further resolves the early diversification of eudicots”. Proc. Natl. Acad. Sci. U.S.A. 107 (10): 4623–8. Bibcode:2010PNAS..107.4623M. doi:10.1073/pnas.0907801107. PMC 2842043. PMID 20176954.
- ^ “Index of /refseq/release/plastid”. ftp.ncbi.nlm.nih.gov. Truy cập ngày 8 tháng 1 năm 2017.
- ^ Wickett NJ, Zhang Y, Hansen SK, Roper JM, Kuehl JV, Plock SA, Wolf PG, DePamphilis CW, Boore JL, Goffinet B (tháng 2 năm 2008). “Functional gene losses occur with minimal size reduction in the plastid genome of the parasitic liverwort Aneura mirabilis”. Mol. Biol. Evol. 25 (2): 393–401. doi:10.1093/molbev/msm267. PMID 18056074.
- ^ Plastid genome evolution of the non-photosynthetic liverwort Aneura mirabilis (Malmb.) Wickett & Goffinet (Aneuraceae)
- ^ Masanori Kugita; Akira Kaneko, Yuhei Yamamoto, Yuko Takeya, Tohoru Matsumoto and Koichi Yoshinaga (1986). “The complete nucleotide sequence of the hornwort (Anthoceros formosae) chloroplast genome: insight into the earliest land plants”. Nucleic Acids Research. 31 (2): 572–4. doi:10.1093/nar/gkg155.Quản lý CS1: nhiều tên: danh sách tác giả (liên kết)
- ^ K Ohyama, Fukuzawa, H., Kohchi, T., Shirai, H., Sano, T., Chang Z, Aota S, Inokuchi H, Ozeki H (2003). “Chloroplast gene organization deduced from complete sequence of liverwort Marchantia polymorpha chloroplast DNA”. Nature. 322 (6079): 716–721. Bibcode:1986Natur.322..572O. doi:10.1038/322572a0.
- ^ Juan Carlos Villarreal; Laura Lowe Forrest, Norman Wickett and Bernard Goffinet (2013). “The plastid genome of the hornwort Nothoceros aenigmaticus (Dendrocerotaceae): Phylogenetic signal in inverted repeat expansion, pseudogenization, and intron gain” (PDF). Am.J.Bot. 103 (3): 467–477. doi:10.3732/ajb.1200429.
- ^ Christopher Grosche; Helena T. Funk, Uwe G. Maier and Stefan Zauner (2012). “The Chloroplast Genome of Pellia endiviifolia: Gene Content, RNA-Editing Pattern, and the Origin of Chloroplast Editing” (PDF). Genome Biology and Evolution. 4 (12): 1349–1357. doi:10.1093/gbe/evs114.
- ^ Sugiura C, Kobayashi Y, Aoki S, Sugita C, Sugita M (2003). “Complete chloroplast DNA sequence of the moss Physcomitrella patens: evidence for the loss and relocation of rpoA from the chloroplast to the nucleus”. Nucleic Acids Res. 31 (18): 5324–31. doi:10.1093/nar/gkg726. PMC 203311. PMID 12954768.
- ^ Laura L. Forrest; Norman J. Wickett, Cymon J. Cox & Bernard Goffinet (2011). “Deep sequencing of Ptilidium (Ptilidiaceae) suggests evolutionary stasis in liverwort plastid genome structure” (PDF). Plant Ecology and Evolution. 144 (1): 29–43.
- ^ Melvin J Olive; Andrew G Murdock, Brent D Mishler, Jennifer V Kuehl, Jeffrey L Boore, Dina F Mandoli, Karin DE Everett, Paul G Wolf, Aaron M Duffy and Kenneth G Karol (2010). “Chloroplast genome sequence of the moss Tortula ruralis: Gene content, polymorphism, and structural arrangement relative to other green plant chloroplast genomes”. BMC Genomics. 11: 143. doi:10.1186/1471-2164-11-143.Quản lý CS1: nhiều tên: danh sách tác giả (liên kết)
- ^ Wolf PG, Rowe CA, Sinclair RB, Hasebe M (2003). “Complete nucleotide sequence of the chloroplast genome from a leptosporangiate fern, Adiantum capillus-veneris L”. DNA Res. 10 (2): 59–65. doi:10.1093/dnares/10.2.59. PMID 12755170.
- ^ Gao L, Yi X, Yang YX, Su YJ, Wang T (2009). “Complete chloroplast genome sequence of a tree fern Alsophila spinulosa: insights into evolutionary changes in fern chloroplast genomes”. BMC Evolutionary Biology. 9: 130. doi:10.1186/1471-2148-9-130.
- ^ Roper, Jessie M.; Kellon Hansen, S.; Wolf, Paul G.; Karol, Kenneth G.; Mandoli, Dina F.; Everett, Karin D. E.; Kuehl, Jennifer; Boore, Jeffrey L. (2007). “The Complete Plastid Genome Sequence of Angiopteris evecta (G. Forst.) Hoffm. (Marattiaceae)”. American Fern Journal. 97 (2): 95–106. doi:10.1640/0002-8444(2007)97[95:TCPGSO]2.0.CO;2.
- ^ Wolf PG, Karol KG, Mandoli DF, Kuehl J, Arumuganathan K, Ellis MW, Mishler BD, Kelch DG, Olmstead RG, Boore JL (2005). “The first complete chloroplast genome sequence of a lycophyte, Huperzia lucidula (Lycopodiaceae)”. Gene. 350 (2): 117–28. doi:10.1016/j.gene.2005.01.018. PMID 15788152.
- ^ Tatsuya Wakasugi, A. Nishikawa, Kyoji Yamada, and Masahiro Sugiura. 1998. "Complete nucleotide sequence of the plastid genome from a fern, Psilotum nudum". Endocytobiology and Cell Research 13(supplement):147. see External links below.
- ^ Tsuji S, Ueda K, Nishiyama T, Hasebe M, Yoshikawa S, Konagaya A, Nishiuchi T, Yamaguchi K (2007). “The chloroplast genome from a lycophyte (microphyllophyte), Selaginella uncinata, has a unique inversion, transpositions and many gene losses”. J. Plant Res. 120 (2): 281–90. doi:10.1007/s10265-006-0055-y. PMID 17297557.
- ^ Hirao T, Watanabe A, Kurita M, Kondo T, Takata K (2008). “Complete nucleotide sequence of the Cryptomeria japonica D. Don. chloroplast genome and comparative chloroplast genomics: diversified genomic structure of coniferous species”. BMC Plant Biol. 8: 70. doi:10.1186/1471-2229-8-70. PMC 2443145. PMID 18570682.
- ^ a b c d e f Jansen RK, Cai Z, Raubeson LA, Daniell H, Depamphilis CW, Leebens-Mack J, Müller KF, Guisinger-Bellian M, Haberle RC, Hansen AK, Chumley TW, Lee SB, Peery R, McNeal JR, Kuehl JV, Boore JL (2007). “Analysis of 81 genes from 64 plastid genomes resolves relationships in angiosperms and identifies genome-scale evolutionary patterns”. Proc. Natl. Acad. Sci. U.S.A. 104 (49): 19369–74. Bibcode:2007PNAS..10419369J. doi:10.1073/pnas.0709121104. PMC 2148296. PMID 18048330.
- ^ Wu CS, Wang YN, Liu SM, Chaw SM (2007). “Chloroplast genome (cpDNA) of Cycas taitungensis and 56 cp protein-coding genes of Gnetum parvifolium: insights into cpDNA evolution and phylogeny of extant seed plants”. Mol. Biol. Evol. 24 (6): 1366–79. doi:10.1093/molbev/msm059. PMID 17383970.
- ^ a b Leebens-Mack J, Raubeson LA, Cui L, Kuehl JV, Fourcade MH, Chumley TW, Boore JL, Jansen RK, depamphilis CW (2005). “Identifying the basal angiosperm node in chloroplast genome phylogenies: sampling one's way out of the Felsenstein zone”. Mol. Biol. Evol. 22 (10): 1948–63. doi:10.1093/molbev/msi191. PMID 15944438.
- ^ Wakasugi T, Tsudzuki J, Ito S, Nakashima K, Tsudzuki T, Sugiura M (1994). “Loss of all ndh genes as determined by sequencing the entire chloroplast genome of the black pine Pinus thunbergii”. Proc. Natl. Acad. Sci. U.S.A. 91 (21): 9794–8. Bibcode:1994PNAS...91.9794W. doi:10.1073/pnas.91.21.9794. PMC 44903. PMID 7937893.
- ^ McCoy SR, Kuehl JV, Boore JL, Raubeson LA (2008). “The complete plastid genome sequence of Welwitschia mirabilis: an unusually compact plastome with accelerated divergence rates”. BMC Evol. Biol. 8: 130. doi:10.1186/1471-2148-8-130. PMC 2386820. PMID 18452621.
- ^ a b c d e f g h i j Guisinger et al, Implications of the Plastid Genome Sequence of Typha (Typhaceae, Poales) for Understanding Genome Evolution in Poaceae, J Mol Evol 70: 149–166 (2010)
- ^ Yan, Minghui; Moore, Michael J.; Meng, Aiping; Yao, Xiaohong; Wang, Hengchang (ngày 21 tháng 9 năm 2016). “The first complete plastome sequence of the basal asterid family Styracaceae (Ericales) reveals a large inversion”. Plant Systematics and Evolution (bằng tiếng Anh). 303 (1): 61–70. doi:10.1007/s00606-016-1352-0. ISSN 0378-2697.
- ^ a b Kim, Jung Sung; Kim, Joo-Hwan (ngày 18 tháng 6 năm 2013). “Comparative Genome Analysis and Phylogenetic Relationship of Order Liliales Insight from the Complete Plastid Genome Sequences of Two Lilies (Lilium longiflorum and Alstroemeria aurea)”. PLOS ONE. 8 (6): e68180. doi:10.1371/journal.pone.0068180. ISSN 1932-6203. PMC 3688979. PMID 23950788.
- ^ W Goremykin; Hirsch-Ernst KI; Wolfl S; Hellwig FH (2003). “Analysis of the Amborella trichopoda chloroplast genome sequence suggests that Amborella is not a basal angiosperm”. Mol Bio Evol. 20: 1445–1454.
- ^ Sato S, Nakamura Y, Kaneko T, Asamizu E, Tabata S (1999). “Complete structure of the chloroplast genome of Arabidopsis thaliana”. DNA Res. 6 (5): 283–90. doi:10.1093/dnares/6.5.283. PMID 10574454.
- ^ Schmitz-Linneweber C, Regel R, Du TG, Hupfer H, Herrmann RG, Maier RM (2002). “The plastid chromosome of Atropa belladonna and its comparison with that of Nicotiana tabacum: the role of RNA editing in generating divergence in the process of plant speciation”. Mol. Biol. Evol. 19 (9): 1602–12. doi:10.1093/oxfordjournals.molbev.a004222. PMID 12200487.
- ^ a b c d Hansen DR, Dastidar SG, Cai Z, Penaflor C, Kuehl JV, Boore JL, Jansen RK (2007). “Phylogenetic and evolutionary implications of complete chloroplast genome sequences of four early-diverging angiosperms: Buxus (Buxaceae), Chloranthus (Chloranthaceae), Dioscorea (Dioscoreaceae), and Illicium (Schisandraceae)”. Mol. Phylogenet. Evol. 45 (2): 547–63. doi:10.1016/j.ympev.2007.06.004. PMID 17644003.
- ^ Goremykin, V.; Hirsch-Ernst, K. I.; Lfl, S.; Hellwig, F. H. (2003). “The chloroplast genome of the basal angiosperm Calycanthus fertilis – structural and phylogenetic analyses”. Plant Systematics and Evolution. 242 (1–4): 119–135. doi:10.1007/s00606-003-0056-4.
- ^ Yang, Yongzhi; Wang, Mingcheng; Lu, Zhiqiang; Xie, Xiuyue; Feng, Shuo (ngày 4 tháng 1 năm 2017). “Characterization of the complete chloroplast genome of Carpinus tientaiensis”. Conservation Genetics Resources (bằng tiếng Anh): 1–3. doi:10.1007/s12686-016-0668-y. ISSN 1877-7252.
- ^ Bausher MG, Singh ND, Lee SB, Jansen RK, Daniell H (2006). “The complete chloroplast genome sequence of Citrus sinensis (L.) Osbeck var 'Ridge Pineapple': organization and phylogenetic relationships to other angiosperms”. BMC Plant Biol. 6: 21. doi:10.1186/1471-2229-6-21. PMC 1599732. PMID 17010212.
- ^ Huang, Ya-Yi; Matzke, Antonius J. M.; Matzke, Marjori (ngày 30 tháng 8 năm 2013). “Complete Sequence and Comparative Analysis of the Chloroplast Genome of Coconut Palm (Cocos nucifera)”. PLoS ONE. 8 (8): e74736. Bibcode:2013PLoSO...874736H. doi:10.1371/journal.pone.0074736. ISSN 1932-6203. PMC 3758300. PMID 24023703.
- ^ Samson N, Bausher MG, Lee SB, Jansen RK, Daniell H (2007). “The complete nucleotide sequence of the coffee (Coffea arabica L.) chloroplast genome: organization and implications for biotechnology and phylogenetic relationships amongst angiosperms”. Plant Biotechnol. J. 5 (2): 339–53. doi:10.1111/j.1467-7652.2007.00245.x. PMC 3473179. PMID 17309688.
- ^ Leseberg CH, Duvall MR (2009). “The complete chloroplast genome of Coix lacryma-jobi and a comparative molecular evolutionary analysis of plastomes in cereals”. J. Mol. Evol. 69 (4): 311–8. doi:10.1007/s00239-009-9275-9. PMID 19777151.
- ^ Wicke, Susann; Müller, Kai F.; Pamphilis, Claude W. de; Quandt, Dietmar; Wickett, Norman J.; Zhang, Yan; Renner, Susanne S.; Schneeweiss, Gerald M. (ngày 1 tháng 10 năm 2013). “Mechanisms of Functional and Physical Genome Reduction in Photosynthetic and Nonphotosynthetic Parasitic Plants of the Broomrape Family”. The Plant Cell (bằng tiếng Anh). 25 (10): 3711–3725. doi:10.1105/tpc.113.113373. ISSN 1532-298X. PMC 3877813. PMID 24143802.
- ^ Plader W, Yukawa Y, Sugiura M, Malepszy S (2007). “The complete structure of the cucumber (Cucumis sativus L.) chloroplast genome: its composition and comparative analysis”. Cell. Mol. Biol. Lett. 12 (4): 584–94. doi:10.2478/s11658-007-0029-7. PMID 17607527.
- ^ a b McNeal JR, Kuehl JV, Boore JL, de Pamphilis CW (2007). “Complete plastid genome sequences suggest strong selection for retention of photosynthetic genes in the parasitic plant genus Cuscuta”. BMC Plant Biol. 7: 57. doi:10.1186/1471-2229-7-57. PMC 2216012. PMID 17956636.
- ^ a b Funk HT, Berg S, Krupinska K, Maier UG, Krause K (2007). “Complete DNA sequences of the plastid genomes of two parasitic flowering plant species, Cuscuta reflexa and Cuscuta gronovii”. BMC Plant Biol. 7: 45. doi:10.1186/1471-2229-7-45. PMC 2089061. PMID 17714582.
- ^ Lin, Choun-Sea; Chen, Jeremy J. W.; Huang, Yao-Ting; Chan, Ming-Tsair; Daniell, Henry; Chang, Wan-Jung; Hsu, Chen-Tran; Liao, De-Chih; Wu, Fu-Huei (ngày 12 tháng 3 năm 2015). “The location and translocation of ndh genes of chloroplast origin in the Orchidaceae family”. Scientific Reports (bằng tiếng Anh). 5: 9040. Bibcode:2015NatSR...5E9040L. doi:10.1038/srep09040. ISSN 2045-2322. PMC 4356964. PMID 25761566.
- ^ Roquet, Cristina; Coissac, Éric; Cruaud, Corinne; Boleda, Martí; Boyer, Frédéric; Alberti, Adriana; Gielly, Ludovic; Taberlet, Pierre; Thuiller, Wilfried (ngày 1 tháng 10 năm 2016). “Understanding the evolution of holoparasitic plants: the complete plastid genome of the holoparasite Cytinus hypocistis (Cytinaceae)”. Annals of Botany (bằng tiếng Anh). 118 (5): 885–896. doi:10.1093/aob/mcw135. ISSN 0305-7364. PMC 5055816. PMID 27443299.
- ^ Ruhlman T, Lee SB, Jansen RK, Hostetler JB, Tallon LJ, Town CD, Daniell H (2006). “Complete plastid genome sequence of Daucus carota: implications for biotechnology and phylogeny of angiosperms”. BMC Genomics. 7: 222. doi:10.1186/1471-2164-7-222. PMC 1579219. PMID 16945140.
- ^ a b c Cai Z, Penaflor C, Kuehl JV, Leebens-Mack J, Carlson JE, dePamphilis CW, Boore JL, Jansen RK (2006). “Complete plastid genome sequences of Drimys, Liriodendron, and Piper: implications for the phylogenetic relationships of magnoliids”. BMC Evol. Biol. 6: 77. doi:10.1186/1471-2148-6-77. PMC 1626487. PMID 17020608.
- ^ Wolfe KH, Morden CW, Palmer JD (tháng 11 năm 1992). “Function and evolution of a minimal plastid genome from a nonphotosynthetic parasitic plant”. Proc. Natl. Acad. Sci. U.S.A. 89 (22): 10648–52. Bibcode:1992PNAS...8910648W. doi:10.1073/pnas.89.22.10648. PMC 50398. PMID 1332054.
- ^ a b Schelkunov, Mikhail I.; Shtratnikova, Viktoria Yu; Nuraliev, Maxim S.; Selosse, Marc-Andre; Penin, Aleksey A.; Logacheva, Maria D. (ngày 1 tháng 4 năm 2015). “Exploring the Limits for Reduction of Plastid Genomes: A Case Study of the Mycoheterotrophic Orchids Epipogium aphyllum and Epipogium roseum”. Genome Biology and Evolution (bằng tiếng Anh). 7 (4): 1179–1191. doi:10.1093/gbe/evv019. ISSN 1759-6653. PMC 4419786. PMID 25635040.
- ^ a b Blazier, John C.; Jansen, Robert K.; Mower, Jeffrey P.; Govindu, Madhu; Zhang, Jin; Weng, Mao-Lun; Ruhlman, Tracey A. (ngày 1 tháng 6 năm 2016). “Variable presence of the inverted repeat and plastome stability in Erodium”. Annals of Botany. 117 (7): 1209–1220. doi:10.1093/aob/mcw065. ISSN 0305-7364. PMC 4904181. PMID 27192713.
- ^ a b c Guisinger, Mary M.; Kuehl, Jennifer V.; Boore, Jeffrey L.; Jansen, Robert K. (ngày 1 tháng 1 năm 2011). “Extreme Reconfiguration of Plastid Genomes in the Angiosperm Family Geraniaceae: Rearrangements, Repeats, and Codon Usage”. Molecular Biology and Evolution (bằng tiếng Anh). 28 (1): 583–600. doi:10.1093/molbev/msq229. ISSN 0737-4038. PMID 20805190.
- ^ Steane DA (2005). “Complete nucleotide sequence of the chloroplast genome from the Tasmanian blue gum, Eucalyptus globulus (Myrtaceae)”. DNA Res. 12 (3): 215–20. doi:10.1093/dnares/dsi006. PMID 16303753.
- ^ Logacheva MD, Samigullin TH, Dhingra A, Penin AA (2008). “Comparative chloroplast genomics and phylogenetics of Fagopyrum esculentum ssp. ancestrale -a wild ancestor of cultivated buckwheat”. BMC Plant Biol. 8: 59. doi:10.1186/1471-2229-8-59. PMC 2430205. PMID 18492277.
- ^ Saski C, Lee SB, Daniell H, Wood TC, Tomkins J, Kim HG, Jansen RK (2005). “Complete chloroplast genome sequence of Gycine max and comparative analyses with other legume genomes”. Plant Mol. Biol. 59 (2): 309–22. doi:10.1007/s11103-005-8882-0. PMID 16247559.
- ^ Rashid Ismael Hag Ibrahim; Jun-Ichi Azuma and Masahiro Sakamoto (2006). “Complete Nucleotide Sequence of the Cotton (Gossypium barbadense L.) Chloroplast Genome with a Comparative Analysis of Sequences among 9 Dicot Plants”. Genes & Genetic Systems. 81 (5): 311–321. doi:10.1266/ggs.81.311.[liên kết hỏng]
- ^ Lee SB, Kaittanis C, Jansen RK, Hostetler JB, Tallon LJ, Town CD, Daniell H (2006). “The complete chloroplast genome sequence of Gossypium hirsutum: organization and phylogenetic relationships to other angiosperms”. BMC Genomics. 7: 61. doi:10.1186/1471-2164-7-61. PMC 1513215. PMID 16553962.
- ^ a b Timme RE, Kuehl JV, Boore JL, Jansen RK (2007). “A comparative analysis of the Lactuca and Helianthus (Asteraceae) plastid genomes: identification of divergent regions and categorization of shared repeats”. Am. J. Bot. 94 (3): 302–12. doi:10.3732/ajb.94.3.302. PMID 21636403.
- ^ Naumann, Julia; Der, Joshua P.; Wafula, Eric K.; Jones, Samuel S.; Wagner, Sarah T.; Honaas, Loren A.; Ralph, Paula E.; Bolin, Jay F.; Maass, Erika; Neinhuis, Christoph; Wanke, Stefan; dePamphilis, Claude W. (ngày 1 tháng 2 năm 2016). “Detecting and Characterizing the Highly Divergent Plastid Genome of the Nonphotosynthetic Parasitic Plant Hydnora visseri (Hydnoraceae)”. Genome Biology and Evolution (bằng tiếng Anh). 8 (2): 345–363. doi:10.1093/gbe/evv256. ISSN 1759-6653. PMC 4779604. PMID 26739167.Quản lý CS1: ngày tháng và năm (liên kết)
- ^ Lee HL, Jansen RK, Chumley TW, Kim KJ (2007). “Gene relocations within chloroplast genomes of Jasminum and Menodora (Oleaceae) are due to multiple, overlapping inversions”. Mol. Biol. Evol. 24 (5): 1161–80. doi:10.1093/molbev/msm036. PMID 17329229.
- ^ Hu, Yiheng; Woeste, Keith E.; Zhao, Peng (ngày 1 tháng 1 năm 2017). “Completion of the Chloroplast Genomes of Five Chinese Juglans and Their Contribution to Chloroplast Phylogeny”. Plant Evolution and Development: 1955. doi:10.3389/fpls.2016.01955. PMC 5216037.
- ^ Mardanov AV, Ravin NV, Kuznetsov BB, Samigullin TH, Antonov AS, Kolganova TV, Skyabin KG (2008). “Complete sequence of the duckweed (Lemna minor) chloroplast genome: structural organization and phylogenetic relationships to other angiosperms”. J. Mol. Evol. 66 (6): 555–64. doi:10.1007/s00239-008-9091-7. PMID 18463914.
- ^ Malé, Pierre-Jean G.; Bardon, Léa; Besnard, Guillaume; Coissac, Eric; Delsuc, Frédéric; Engel, Julien; Lhuillier, Emeline; Scotti-Saintagne, Caroline; Tinaut, Alexandra (ngày 1 tháng 9 năm 2014). “Genome skimming by shotgun sequencing helps resolve the phylogeny of a pantropical tree family”. Molecular Ecology Resources (bằng tiếng Anh). 14 (5): 966–975. doi:10.1111/1755-0998.12246. ISSN 1755-0998.
- ^ Liang H, Carlson JE, Leebens-Mack JH, Wall PK, Mueller LA, Buzgo M, Landherr LL, Hu Y, DiLoreto DS, Ilut DC, Field D, Tanksley SD, Ma H, Claude (2008). “An EST database for Liriodendron tulipifera L. floral buds: the first EST resource for functional and comparative genomics in Liriodendron”. Tree Genetics and Genomes. 4 (3): 419–433. doi:10.1007/s11295-007-0120-2.
- ^ Kato T, Kaneko T, Sato S, Nakamura Y, Tabata S (2000). “Complete structure of the chloroplast genome of a legume, Lotus japonicus”. DNA Res. 7 (6): 323–30. doi:10.1093/dnares/7.6.323. PMID 11214967.
- ^ Daniell H, Wurdack KJ, Kanagaraj A, Lee SB, Saski C, Jansen RK (2008). “The complete nucleotide sequence of the cassava (Manihot esculenta) chloroplast genome and the evolution of atpF in Malpighiales: RNA editing and multiple losses of a group II intron”. Theor. Appl. Genet. 116 (5): 723–37. doi:10.1007/s00122-007-0706-y. PMC 2587239. PMID 18214421.
- ^ Ravin, Nikolai V.; Gruzdev, Eugeny V.; Beletsky, Alexey V.; Mazur, Alexander M.; Prokhortchouk, Egor B.; Filyushin, Mikhail A.; Kochieva, Elena Z.; Kadnikov, Vitaly V.; Mardanov, Andrey V. (ngày 1 tháng 1 năm 2016). “The loss of photosynthetic pathways in the plastid and nuclear genomes of the non-photosynthetic mycoheterotrophic eudicot Monotropa hypopitys”. BMC Plant Biology. 16 (Suppl 3): 153–161. doi:10.1186/s12870-016-0929-7. ISSN 1471-2229. PMC 5123295.
- ^ Ravi, V.; Khurana, Jitendra P.; Tyagi, Akhilesh K.; Khurana, Paramjit (2006). “The chloroplast genome of mulberry: complete nucleotide sequence, gene organization and comparative analysis”. Tree Genetics and Genomes. 3 (1): 49–59. doi:10.1007/s11295-006-0051-3.
- ^ Shetty, Santoshkumar M.; Shah, Maria Ulfa Md; Makale, Kavyashree; Mohd-Yusuf, Yusmin; Khalid, Norzulaani; Othman, Rofina Yasmin (ngày 1 tháng 7 năm 2016). “Complete Chloroplast Genome Sequence of Corroborates Structural Heterogeneity of Inverted Repeats in Wild Progenitors of Cultivated Bananas and Plantains”. The Plant Genome (bằng tiếng Anh). 9 (2). doi:10.3835/plantgenome2015.09.0089. ISSN 1940-3372. Bản gốc lưu trữ ngày 18 tháng 1 năm 2017. Truy cập ngày 22 tháng 2 năm 2017.
- ^ a b Moore MJ, Dhingra A, Soltis PS, Shaw R, Farmerie WG, Folta KM, Soltis DE (2006). “Rapid and accurate pyrosequencing of angiosperm plastid genomes”. BMC Plant Biol. 6: 17. doi:10.1186/1471-2229-6-17. PMC 1564139. PMID 16934154.
- ^ Logacheva, Maria D.; Schelkunov, Mikhail I.; Penin, Aleksey A. (ngày 1 tháng 1 năm 2011). “Sequencing and Analysis of Plastid Genome in Mycoheterotrophic Orchid Neottia nidus-avis”. Genome Biology and Evolution (bằng tiếng Anh). 3: 1296–1303. doi:10.1093/gbe/evr102. ISSN 1759-6653. PMC 3228488. PMID 21971517.
- ^ Shinozaki K, Ohme M, Tanaka M, Wakasugi T, Hayashida N, Matsubayashi T, Zaita N, Chunwongse J, Obokata J, Yamaguchi-Shinozaki K, Ohto C, Torazawa K, Meng BY, Sugita M, Deno H, Kamogashira T, Yamada K, Kusuda J, Takaiwa F, Kato A, Tohdoh N, Shimada H, Sugiura M (1986). “The complete nucleotide sequence of the tobacco chloroplast genome: its gene organization and expression”. EMBO J. 5 (9): 2043–2049. PMC 1167080. PMID 16453699.
- ^ a b Raubeson LA, Peery R, Chumley TW, Dziubek C, Fourcade HM, Boore JL, Jansen RK (2007). “Comparative chloroplast genomics: analyses including new sequences from the angiosperms Nuphar advena and Ranunculus macranthus”. BMC Genomics. 8: 174. doi:10.1186/1471-2164-8-174. PMC 1925096. PMID 17573971.
- ^ Goremykin VV, Hirsch-Ernst KI, Wölfl S, Hellwig FH (2004). “The chloroplast genome of Nymphaea alba: whole-genome analyses and the problem of identifying the most basal angiosperm”. Mol. Biol. Evol. 21 (7): 1445–54. doi:10.1093/molbev/msh147. PMID 15084683.
- ^ a b c d e Greiner S, Wang X, Rauwolf U, Silber MV, Mayer K, Meurer J, Haberer G, Herrmann RG (2008). “The complete nucleotide sequences of the five genetically distinct plastid genomes of Oenothera, subsection Oenothera: I. sequence evaluation and plastome evolution”. Nucleic Acids Res. 36 (7): 2366–78. doi:10.1093/nar/gkn081. PMC 2367718. PMID 18299283.
- ^ a b Jun Yu et alii (117 authors). 2005. "The Genomes of Oryza sativa: a history of duplications". PLoS Biology 3(2):e38. Epub 2005 Feb 1.
- ^ Hiratsuka J, Shimada H, Whittier R, Ishibashi T, Sakamoto M, Mori M, Kondo C, Honji Y, Sun CR, Meng BY (1989). “The complete sequence of the rice (Oryza sativa) chloroplast genome: intermolecular recombination between distinct tRNA genes accounts for a major plastid DNA inversion during the evolution of the cereals”. Mol. Gen. Genet. 217 (2–3): 185–94. doi:10.1007/BF02464880. PMID 2770692.
- ^ a b c Petersen, Gitte; Cuenca, Argelia; Seberg, Ole (ngày 1 tháng 9 năm 2015). “Plastome Evolution in Hemiparasitic Mistletoes”. Genome Biology and Evolution (bằng tiếng Anh). 7 (9): 2520–2532. doi:10.1093/gbe/evv165. ISSN 1759-6653. PMC 4607522. PMID 26319577.
- ^ Kim KJ, Lee HL (2004). “Complete chloroplast genome sequences from Korean ginseng (Panax schinseng Nees) and comparative analysis of sequence evolution among 17 vascular plants”. DNA Res. 11 (4): 247–61. doi:10.1093/dnares/11.4.247. PMID 15500250.
- ^ Chumley TW, Palmer JD, Mower JP, Fourcade HM, Calie PJ, Boore JL, Jansen RK (2006). “The complete chloroplast genome sequence of Pelargonium x hortorum: organization and evolution of the largest and most highly rearranged chloroplast genome of land plants”. Mol. Biol. Evol. 23 (11): 2175–90. doi:10.1093/molbev/msl089. PMID 16916942.
- ^ Logacheva, Maria D.; Schelkunov, Mikhail I.; Nuraliev, Maxim S.; Samigullin, Tagir H.; Penin, Aleksey A. (ngày 1 tháng 1 năm 2014). “The Plastid Genome of Mycoheterotrophic Monocot Petrosavia stellaris Exhibits Both Gene Losses and Multiple Rearrangements”. Genome Biology and Evolution (bằng tiếng Anh). 6 (1): 238–246. doi:10.1093/gbe/evu001. ISSN 1759-6653. PMC 3914687. PMID 24398375.
- ^ Chang CC, Lin HC, Lin IP, Chow TY, Chen HH, Chen WH, Cheng CH, Lin CY, Liu SM, Chang CC, Chaw SM (2006). “The chloroplast genome of Phalaenopsis aphrodite (Orchidaceae): comparative analysis of evolutionary rate with that of grasses and its phylogenetic implications”. Mol. Biol. Evol. 23 (2): 279–91. doi:10.1093/molbev/msj029. PMID 16207935.
- ^ Guo X, Castillo-Ramírez S, González V, Bustos P, Fernández-Vázquez JL, Santamaría RI, Arellano J, Cevallos MA, Dávila G (2007). “Rapid evolutionary change of common bean (Phaseolus vulgaris L) plastome, and the genomic diversification of legume chloroplasts”. BMC Genomics. 8: 228. doi:10.1186/1471-2164-8-228. PMC 1940014. PMID 17623083.
- ^ a b Bellot, Sidonie; Renner, Susanne S. (ngày 1 tháng 1 năm 2016). “The Plastomes of Two Species in the Endoparasite Genus Pilostyles (Apodanthaceae) Each Retain Just Five or Six Possibly Functional Genes”. Genome Biology and Evolution (bằng tiếng Anh). 8 (1): 189–201. doi:10.1093/gbe/evv251. ISSN 1759-6653. PMC 4758247. PMID 26660355.
- ^ Okumura S, Sawada M, Park YW, Hayashi T, Shimamura M, Takase H, Tomizawa K (2006). “Transformation of poplar (Populus alba) plastids and expression of foreign proteins in tree chloroplasts”. Transgenic Res. 15 (5): 637–46. doi:10.1007/s11248-006-9009-3. PMID 16952016.
- ^ Delannoy, Etienne; Fujii, Sota; Francs-Small, Catherine Colas des; Brundrett, Mark; Small, Ian (ngày 1 tháng 7 năm 2011). “Rampant Gene Loss in the Underground Orchid Rhizanthella gardneri Highlights Evolutionary Constraints on Plastid Genomes”. Molecular Biology and Evolution (bằng tiếng Anh). 28 (7): 2077–2086. doi:10.1093/molbev/msr028. ISSN 0737-4038. PMC 3112369. PMID 21289370.
- ^ Lam, Vivienne K. Y.; Gomez, Marybel Soto; Graham, Sean W. (ngày 1 tháng 8 năm 2015). “The Highly Reduced Plastome of Mycoheterotrophic Sciaphila (Triuridaceae) Is Colinear with Its Green Relatives and Is under Strong Purifying Selection”. Genome Biology and Evolution (bằng tiếng Anh). 7 (8): 2220–2236. doi:10.1093/gbe/evv134. ISSN 1759-6653. PMC 4558852. PMID 26170229.
- ^ Chung HJ, Jung JD, Park HW, Kim JH, Cha HW, Min SR, Jeong WJ, Liu JR (2006). “The complete chloroplast genome sequences of Solanum tuberosum and comparative analysis with Solanaceae species identified the presence of a 241-bp deletion in cultivated potato chloroplast DNA sequence”. Plant Cell Rep. 25 (12): 1369–79. doi:10.1007/s00299-006-0196-4. PMID 16835751.
- ^ Schmitz-Linneweber C, Maier RM, Alcaraz JP, Cottet A, Herrmann RG, Mache R (2001). “The plastid chromosome of spinach (Spinacia oleracea): complete nucleotide sequence and gene organization”. Plant Mol. Biol. 45 (3): 307–15. doi:10.1023/A:1006478403810. PMID 11292076.
- ^ Haberle RC, Fourcade HM, Boore JL, Jansen RK (2008). “Extensive Rearrangements in the Chloroplast Genome of Trachelium caeruleum Are Associated with Repeats and tRNA Genes”. Journal of Molecular Evolution. 66 (4): 350–361. doi:10.1007/s00239-008-9086-4.
- ^ Zhengqiu Cai et al, Extensive Reorganization of the Plastid Genome of Trifolium subterraneum (Fabaceae) Is Associated with Numerous Repeated Sequences and Novel DNA Insertions, J Mol Evol 67: 696–704 (2008)doi:10.1007/s00239-008-9180-7
- ^ Ogihara, Yasunari; Isono, Kazuriho; Kojima, Toshio; Endo, Akira; Hanaoka, Mitsumasa; Shiina, Takashi; Terachi, Toru; Utsugi, Shigeko; và đồng nghiệp (2000). “Chinese Spring Wheat (Triticum aestivum L.) Chloroplast Genome: Complete Sequence and Contig Clones”. Plant Molecular Biology Reporter. 18 (3): 243–253. doi:10.1007/BF02823995.
- ^ Ogihara Y, Isono K, Kojima T, Endo A, Hanaoka M, Shiina T, Terachi T, Utsugi S, Murata M, Mori N, Takumi S, Ikeo K, Gojobori T, Murai R, Murai K, Matsuoka Y, Ohnishi Y, Tajiri H, Tsunewaki K (2002). “Structural features of a wheat plastome as revealed by complete sequencing of chloroplast DNA”. Mol. Genet. Genomics. 266 (5): 740–6. doi:10.1007/s00438-001-0606-9. PMID 11810247.
- ^ Fajardo D, Senalik D, Ames M, Zhu H, Steffan SA, Harbut R, Polashock J, Vorsa N, Gillespie E, Kron K, Zalapa JE (2013). “Complete plastid genome sequence of Vaccinium macrocarpon: structure, gene content, and rearrangements revealed by next generation sequencing”. Tree Genetics & Genomes. 9 (2): 489–498. doi:10.1007/s11295-012-0573-9.
- ^ Jansen RK, Kaittanis C, Saski C, Lee SB, Tomkins J, Alverson AJ, Daniell H (2006). “Phylogenetic analyses of Vitis (Vitaceae) based on complete chloroplast genome sequences: effects of taxon sampling and phylogenetic methods on resolving relationships among rosids”. BMC Evol. Biol. 6: 32. doi:10.1186/1471-2148-6-32. PMC 1479384. PMID 16603088.
- ^ Maier RM, Neckermann K, Igloi GL, Kössel H (1995). “Complete sequence of the maize chloroplast genome: gene content, hotspots of divergence and fine tuning of genetic information by transcript editing”. J. Mol. Biol. 251 (5): 614–28. doi:10.1006/jmbi.1995.0460. PMID 7666415.
- ^ Moore MJ, Bell CD, Soltis PS, Soltis DE (2007). “Using plastid genome-scale data to resolve enigmatic relationships among basal angiosperms”. Proc. Natl. Acad. Sci. U.S.A. 104 (49): 19363–8. Bibcode:2007PNAS..10419363M. doi:10.1073/pnas.0708072104. PMC 2148295. PMID 18048334.
- ^ Gerald A. Tuskan, et alii (110 authors). 2006. "The genome of Black Cottonwood, Populus trichocarpa (Torr. & Gray)". Science 313 (5793):1596-1604.
- ^ “A revised classification of Santalales”. Taxon. 59 (2): 538–558. 2010.
- ^ a b Leliaert, Frederik; Lopez-Bautista, Juan M (2015). “The chloroplast genomes of Bryopsis plumosa and Tydemania expeditionis (Bryopsidales, Chlorophyta): compact genomes and genes of bacterial origin”. BMC Genomics. 16 (1): 204. doi:10.1186/s12864-015-1418-3. ISSN 1471-2164.
- ^ Turmel M, Otis C, Lemieux C (tháng 8 năm 2002). “The chloroplast and mitochondrial genome sequences of the charophyte Chaetosphaeridium globosum: insights into the timing of the events that restructured organelle DNAs within the green algal lineage that led to land plants”. Proc. Natl. Acad. Sci. U.S.A. 99 (17): 11275–80. Bibcode:2002PNAS...9911275T. doi:10.1073/pnas.162203299. PMC 123247. PMID 12161560.
- ^ Wakasugi T, Nagai T, Kapoor M, Sugita M, Ito M, Ito S, Tsudzuki J, Nakashima K, Tsudzuki T, Suzuki Y, Hamada A, Ohta T, Inamura A, Yoshinaga K, Sugiura M (tháng 5 năm 1997). “Complete nucleotide sequence of the chloroplast genome from the green alga Chlorella vulgaris: the existence of genes possibly involved in chloroplast division”. Proc. Natl. Acad. Sci. U.S.A. 94 (11): 5967–72. Bibcode:1997PNAS...94.5967W. doi:10.1073/pnas.94.11.5967. PMC 20890. PMID 9159184.
- ^ Turmel M, Otis C, Lemieux C (2007). “An unexpectedly large and loosely packed mitochondrial genome in the charophycean green alga Chlorokybus atmophyticus”. BMC Genomics. 8: 137. doi:10.1186/1471-2164-8-137. PMC 1894977. PMID 17537252.
- ^ Smith DR, và đồng nghiệp (2010). “The Dunaliella salina organelle genomes: large sequences, inflated with intronic and intergenic DNA”. BMC Plant Biol. 10: 83. doi:10.1186/1471-2229-10-83. PMC 3017802. PMID 20459666.
- ^ de Koning AP, Keeling PJ (2006). “The complete plastid genome sequence of the parasitic green alga Helicosporidium sp. is highly reduced and structured”. BMC Biol. 4: 12. doi:10.1186/1741-7007-4-12. PMC 1463013. PMID 16630350.
- ^ de Cambiaire JC, Otis C, Turmel M, Lemieux C (2007). “The chloroplast genome sequence of the green alga Leptosira terrestris: multiple losses of the inverted repeat and extensive genome rearrangements within the Trebouxiophyceae”. BMC Genomics. 8: 213. doi:10.1186/1471-2164-8-213. PMC 1931444. PMID 17610731.
- ^ a b c Turmel M, Gagnon MC, O'Kelly CJ, Otis C, Lemieux C (tháng 3 năm 2009). “The chloroplast genomes of the green algae Pyramimonas, Monomastix, and Pycnococcus shed new light on the evolutionary history of prasinophytes and the origin of the secondary chloroplasts of euglenids”. Mol. Biol. Evol. 26 (3): 631–48. doi:10.1093/molbev/msn285. PMID 19074760.
- ^ Turmel M, Otis C, Lemieux C (tháng 8 năm 1999). “The complete chloroplast DNA sequence of the green alga Nephroselmis olivacea: insights into the architecture of ancestral chloroplast genomes”. Proc. Natl. Acad. Sci. U.S.A. 96 (18): 10248–53. Bibcode:1999PNAS...9610248T. doi:10.1073/pnas.96.18.10248. PMC 17874. PMID 10468594.
- ^ Brouard JS, Otis C, Lemieux C, Turmel M (2008). “Chloroplast DNA sequence of the green alga Oedogonium cardiacum (Chlorophyceae): unique genome architecture, derived characters shared with the Chaetophorales and novel genes acquired through horizontal transfer”. BMC Genomics. 9: 290. doi:10.1186/1471-2164-9-290. PMC 2442088. PMID 18558012.
- ^ Pombert JF, Lemieux C, Turmel M (2006). “The complete chloroplast DNA sequence of the green alga Oltmannsiellopsis viridis reveals a distinctive quadripartite architecture in the chloroplast genome of early diverging ulvophytes”. BMC Biol. 4: 3. doi:10.1186/1741-7007-4-3. PMC 1402334. PMID 16472375.
- ^ Robbens S, Derelle E, Ferraz C, Wuyts J, Moreau H, Van de Peer Y (tháng 4 năm 2007). “The complete chloroplast and mitochondrial DNA sequence of Ostreococcus tauri: organelle genomes of the smallest eukaryote are examples of compaction”. Mol. Biol. Evol. 24 (4): 956–68. doi:10.1093/molbev/msm012. PMID 17251180.
- ^ Pombert JF, Otis C, Lemieux C, Turmel M (tháng 9 năm 2005). “The chloroplast genome sequence of the green alga Pseudendoclonium akinetum (Ulvophyceae) reveals unusual structural features and new insights into the branching order of chlorophyte lineages”. Mol. Biol. Evol. 22 (9): 1903–18. doi:10.1093/molbev/msi182. PMID 15930151.
- ^ de Cambiaire JC, Otis C, Lemieux C, Turmel M (2006). “The complete chloroplast genome sequence of the chlorophycean green alga Scenedesmus obliquus reveals a compact gene organization and a biased distribution of genes on the two DNA strands”. BMC Evol. Biol. 6: 37. doi:10.1186/1471-2148-6-37. PMC 1513399. PMID 16638149.
- ^ Turmel M, Otis C, Lemieux C (2005). “The complete chloroplast DNA sequences of the charophycean green algae Staurastrum and Zygnema reveal that the chloroplast genome underwent extensive changes during the evolution of the Zygnematales”. BMC Biol. 3: 22. doi:10.1186/1741-7007-3-22. PMC 1277820. PMID 16236178.
- ^ Bélanger AS, Brouard JS, Charlebois P, Otis C, Lemieux C, Turmel M (tháng 11 năm 2006). “Distinctive architecture of the chloroplast genome in the chlorophycean green alga Stigeoclonium helveticum”. Mol. Genet. Genomics. 276 (5): 464–77. doi:10.1007/s00438-006-0156-2. PMID 16944205.[liên kết hỏng]
- ^ Melton, James T.; Leliaert, Frederik; Tronholm, Ana; Lopez-Bautista, Juan M. (2015). “The Complete Chloroplast and Mitochondrial Genomes of the Green Macroalga Ulva sp. UNA00071828 (Ulvophyceae, Chlorophyta)”. PLoS ONE. 10 (4): e0121020. Bibcode:2015PLoSO..1021020M. doi:10.1371/journal.pone.0121020. ISSN 1932-6203. PMC 4388391. PMID 25849557.
- ^ Smith DR, Lee RW (tháng 3 năm 2009). “The mitochondrial and plastid genomes of Volvox carteri: bloated molecules rich in repetitive DNA”. BMC Genomics. 10 (132): 132. doi:10.1186/1471-2164-10-132. PMC 2670323. PMID 19323823.
- ^ Ohta N, Matsuzaki M, Misumi O, Miyagishima SY, Nozaki H, Tanaka K, Shin-I T, Kohara Y, Kuroiwa T (2003). “Complete sequence and analysis of the plastid genome of the unicellular red alga Cyanidioschyzon merolae”. DNA Res. 10 (2): 67–77. doi:10.1093/dnares/10.2.67. PMID 12755171.
- ^ Glöckner G, Rosenthal A, Valentin K (2000). “The structure and gene repertoire of an ancient red algal plastid genome”. J. Mol. Evol. 51 (4): 382–90. doi:10.1007/s002390010101. PMID 11040290.[liên kết hỏng]
- ^ Hagopian JC, Reis M, Kitajima JP, Bhattacharya D, de Oliveira MC (2004). “Comparative analysis of the complete plastid genome sequence of the red alga Gracilaria tenuistipitata var. liui provides insights into the evolution of rhodoplasts and their relationship to other plastids”. J. Mol. Evol. 59 (4): 464–77. doi:10.1007/s00239-004-2638-3. PMID 15638458.
- ^ Douglas SE, Penny SL (1999). “The plastid genome of the cryptophyte alga, Guillardia theta: complete sequence and conserved synteny groups confirm its common ancestry with red algae”. J. Mol. Evol. 48 (2): 236–44. doi:10.1007/PL00006462. PMID 9929392.[liên kết hỏng]
- ^ E. Virginia Armbrust, et alii (42 authors). 2004. "The Genome of the Diatom Thalassiosira pseudonana: Ecology, Evolution, and Metabolism". Science 306(5693):79-86.
- ^ Rogers MB, Gilson PR, Su V, McFadden GI, Keeling PJ (2007). “The complete chloroplast genome of the chlorarachniophyte Bigelowiella natans: evidence for independent origins of chlorarachniophyte and euglenid secondary endosymbionts”. Mol. Biol. Evol. 24 (1): 54–62. doi:10.1093/molbev/msl129. PMID 16990439.
- ^ Hallick RB, Hong L, Drager RG, Favreau MR, Monfort A, Orsat B, Spielmann A, Stutz E (1993). “Complete sequence of Euglena gracilis chloroplast DNA”. Nucleic Acids Res. 21 (15): 3537–44. doi:10.1093/nar/21.15.3537. PMC 331456. PMID 8346031.
- ^ Cai X, Fuller AL, McDougald LR, Zhu G (2003). “Apicoplast genome of the coccidian Eimeria tenella”. Gene. 321: 39–46. doi:10.1016/j.gene.2003.08.008. PMID 14636990.
- ^ Nowack EC, Melkonian M, Glöckner G (2008). “Chromatophore genome sequence of Paulinella sheds light on acquisition of photosynthesis by eukaryotes”. Curr. Biol. 18 (6): 410–8. doi:10.1016/j.cub.2008.02.051. PMID 18356055.
Liên kết ngoài sửa
- HAMAP proteomes: Plastids Lưu trữ 2006-10-04 tại Archive.today
- Complete Plastid Genomes
- Chloroplast Genome Database Lưu trữ 2011-07-20 tại Wayback Machine
- NCBI Eukaryotic Plastid Genomes
- chloroplast genome list Lưu trữ 2012-04-20 tại Wayback Machine In: Montreal genomics Lưu trữ 2012-04-20 tại Wayback Machine
- Psilotum nudum